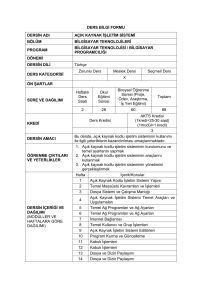
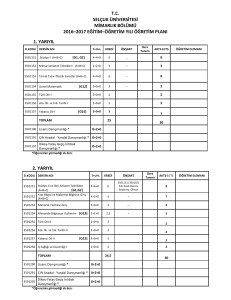
Lamiaceae - Ulusal Tez Merkezi
advertisement

17 T.C. ERCİYES ÜNİVERSİTESİ FEN BİLİMLERİ ENSTİTÜSÜ BİYOLOJİ ANABİLİM DALI PHLOMIS L. (Lamiaceae) CİNSİNE AİT TÜRLER ARASINDAKİ DOĞAL MELEZLERİN ISSR YÖNTEMİYLE İNCELENMESİ Hazırlayan Özge FIRAT Danışman Doç. Dr. Ertuğrul YÜZBAŞIOĞLU Prof. Dr. Mehmet Yaşar DADANDI Yüksek Lisans Tezi Ocak 2016 KAYSERİ 18 T.C. ERCİYES ÜNİVERSİTESİ FEN BİLİMLERİ ENSTİTÜSÜ BİYOLOJİ ANABİLİM DALI PHLOMIS L. (Lamiaceae) CİNSİNE AİT TÜRLER ARASINDAKİ DOĞAL MELEZLERİN ISSR YÖNTEMİYLE İNCELENMESİ (Yüksek Lisans Tezi) Hazırlayan Özge FIRAT Danışman Doç. Dr. Ertuğrul YÜZBAŞIOĞLU Prof. Dr. Mehmet Yaşar DADANDI Bu çalışma; Erciyes Üniversitesi Bilimsel Araştırma Projeleri Birimi Tarafından FBY-11-3423 kodlu proje ile desteklenmiştir. Ocak 2016 KAYSERİ iv TEŞEKKÜR Çalışmalarım boyunca farklı bakış açıları ve bilimsel katkılarıyla beni aydınlatan, yakın ilgi ve yardımlarını esirgemeyen ve bu günlere gelmemde en büyük katkı sahibi sayın hocam Doç. Dr Ertuğrul YÜZBAŞIOĞLU’na, kullanılan bitki materyallerini temin eden sayın hocam Prof. Dr. Mehmet Yaşar DADANDI’ya teşekkürü bir borç bilirim. Deneysel çalışmalarım sırasında karşılaştığım zorlukları aşmamda yardımlarından dolayı Doç. Dr. Fatma ÖZTÜRK KÜP’e ve aynı laboratuvarı paylaştığımız çalışma arkadaşım Fadime ÖZTOPRAK’a teşekkür ederim. Bu tez çalışmasına maddi destek veren Erciyes Üniversitesi Bilimsel Araştırma Projeleri Birimi’ne (Proje No: FBY-11-3423) teşekkür ederim. Ayrıca; çalışmalarım süresince sabır göstererek beni daima destekleyen eşime ve aileme en içten teşekkürlerimi sunarım. Özge FIRAT Kayseri, Ocak 2016 v PHLOMIS L. (Lamiaceae) CİNSİNE AİT TÜRLER ARASINDAKİ DOĞAL MELEZLERİN ISSR YÖNTEMİYLE İNCELENMESİ Özge FIRAT Erciyes Üniversitesi, Fen Bilimleri Enstitüsü Yüksek Lisans Tezi, Ocak 2016 Tez Danışmanları: Doç. Dr. Ertuğrul YÜZBAŞIOĞLU Prof. Dr. Mehmet Yaşar DADANDI ÖZET Bu araştırmada 41 Phlomis taksonu, 1 Phlomoides taksonu ve Phlomis taksonları arasında oluşan 36 adet doğal Phlomis melezine ait 193 örnek kullanılarak, doğal melezleşme ve türler arası ilişki ISSR yöntemi ve 24 farklı morfolojik karakter bakımından incelenmiştir. İzole edilen genomik DNA’dan ISSR bölgesine yönelik 17 farklı primer ile amplifikasyon gerçekleştirilmiştir. Morfolojik ve moleküler verilere dayalı filogram ağaçları NTYS programı kullanılarak; filogenetik network grafikleri Splittree programı kullanılarak oluşturulmuştur. ISSR ve morfolojik verilerden elde edilen filogram ağaçları incelendiğinde her ikisinde de 193 Phlomis örneği iki ana gruba ayrılmaktadır. Fakat bu gruplanma Phlomis cinsinin Phlomis ve Phlomoides seksiyon ayırımını ve Phlomis seksiyonunun Dendrophlomis, Oxyphlomis ve Gymnophlomis olmak üzere 3 alt seksiyona ayırımını da desteklememektedir. Morfolojik verilerden elde edilen filogram ağacında 46 melez örneğinden 37’si atalarının dışındaki türler ve melezlerle bir arada gruplanmışken ISSR ağacında ise 46 melez örneğinden 21’i atalarının dışındaki türler ve melezlerle bir arada gruplanmıştır. Morfolojik verilerden elde edilen network grafiği 193 Phlomis örneğini iki ana gruba ayırmakta iken ISSR yöntemi 10 gruba ayırmaktadır. Morfolojik veriden elde edilen network grafiğinde 46 melez örneğinin 37’si ya atalarının birinin hemen yanında ya da atalarının bulunduğu grup içerisinde yer almıştır. ISSR verisinden elde edilen network grafiğinde ise 46 melez örneğinin 40 tanesi ya atalarının birinin hemen yanında ya da atalarının en azından birinin bulunduğu grup içerisinde yer almıştır. Anahtar Kelimeler: Phlomis, Lamiacea, ISSR, melezleşme vi ISSR ANALYSIS OF NATURAL HYBRIDIS BETWEEN THE SPECIES OF THE GENUS PHLOMIS L. (Lamiaceae) Özge FIRAT Erciyes Universty, Institute of Science MSc. Thesis, january 2016 Supervisors: Assoc. Prof. Dr. Ertuğrul YÜZBAŞIOĞLU, Prof. Dr. Mehmet Yaşar DADANDI ABSTRACT In this study, 193 samples from 41 Phlomis taxa, one Phlomoides taxon and 36 natural Phlomis hybrids were used to assess natural hybridization and phylogenetic relationships in the genus Phylomis by using ISSR method and 24 different morphological characters. ISSR regions has been amplified from the genomic DNAs using 17 different ISSR primers. NTYS and Splittree package programmes were used to build phylogenetic trees and phylogenetic network graphics. When the phylogram trees obtained from ISSR method and morphological data were exaimed, 193 samples of the genus Phlomis were divided into two groups. However, this grouping does not support both the sectional division of the genus Phlomis as Phlomoides and Phlomis and also the subsectional division of the section Phlomis as Dendrophlomis, Oxyphlomis and Gymnophlomis. Of 46 Phlomis hybrids, 37 were groupped with non-parental species/other hybrids in the phylogram tree obtained from the morphological data. Similarly, of 46 Phlomis hybrids, 21 were groupped with non-parental species/other hybrids in the phylogram tree obtained from the ISSR data. While the network tree obtained from the morphological data divided the 193 Phlomis samples into two main groups, the network tree obtained from the ISSR data divided the 193 Phlomis samples into 10 groups. In the network tree obtained from the morphological data, , 37 of the 46 Phlomis hybrids were placed either next to the one of their parents or within the groups including at least one of their parents. Similarly, in the network tree obtained from the ISSR data, 40 of the 46 Phlomis hybrids were placed either next to the one of their parents or within the groups including at least one of their parents. Key Words: Phlomis, Lamiaceae, ISSR, hybridisation vii İÇİNDEKİLER PHLOMIS L. (Lamiaceae) CİNSİNE AİT TÜRLER ARASINDAKİ DOĞAL MELEZLERİN ISSR YÖNTEMİYLE İNCELENMESİ BİLİMSEL ETİĞE UYGUNLUK.................................................................................. i YÖNERGEYE UYGUNLUK ....................................................................................... ii ONAY ......................................................................................................................... iii TEŞEKKÜR ................................................................................................................ iv ÖZET ........................................................................................................................... v ABSTRACT ................................................................................................................ vi İÇİNDEKİLER ........................................................................................................... vii KISALTMALAR .......................................................................................................... x TABLOLAR LİSTESİ ................................................................................................. xi ŞEKİLLER LİSTESİ .................................................................................................. xii GİRİŞ .......................................................................................................................... 1 1. BÖLÜM GENEL BİLGİLER ve LİTERATÜR TARAMASI 1.1. Lamiaceae Familyasının Morfolojik Özellikleri .................................................. 6 1.2. Phlomis L. Cinsinin Morfolojik Özellikleri ......................................................... 6 1.2.1. Phlomis L. Cinsinin Sistematikteki Yeri ...................................................... 7 1.2.2. Phlomis L. Cinsi ve Dünya Üzerindeki Yayılışı ........................................... 7 1.2.3. Phlomis L. Cinsi Üzerine Yapılan Çalışmalar ............................................. 8 1.3. Bitkiler Aleminde Melezleşme ........................................................................... 10 1.4. Moleküler Sistematik ......................................................................................... 13 1.4.1. Morfolojik Markırlar ................................................................................. 13 1.4.2. Biyokimyasal Markırlar ............................................................................. 14 1.4.3. Moleküler Markırlar .................................................................................. 14 1.4.3.1. Restriksiyon Parça Uzunluk Polimorfizmi - Restriction Fragment Lenght Polymorphism (RFLP) .......................................................... 15 viii 1.4.3.2. Çoğaltılmış Parça Uzunluk Polimorfizmi - Amplified Fragment Lenght Polymorphism (AFLP) .......................................................... 15 1.4.3.3. Rastgele Çoğaltılmış Polimorfik DNA - Randomly Amplified Polymorphic DNA(s) (RAPD) ............................................................ 16 1.4.3.4. DNA Dizisi Belirlenerek Çoğaltılmış Polimorfik Bölgeler - Sequence Characterized Amplified Regions (SCAR)........................................ 16 1.4.3.5. İçsel Transkribe Edilen Bölge - Internal Transcribed Spacer (ITS) 17 1.4.3.6. Kloroplast DNA (cpDNA)................................................................... 17 1.4.3.7. Basit Dizi Tekrarları (Mikrosatallitler) - Simple Sequence Repeats (SSR) ................................................................................................... 17 1.4.3.8. Basit Dizi Tekrarları Arası - Inter Simple Sequence Repeats (ISSR) ................................................................................................. 18 2. BÖLÜM MATERYAL VE METOD 2.1. Materyal ............................................................................................................. 21 2.1.1. Bitki Materyali ........................................................................................... 21 2.1.2. Kullanılan Kimyasal Maddeler ve Çözeltiler ............................................ 31 2.1.2.1. DNA İzolasyonunda Kullanılan Kimyasal Maddeler ve Çözeltiler .. 31 2.1.2.2. PCR Reaksiyonlarında Kullanılan Kimyasal Maddeler ve Çözeltiler............................................................................................. 32 2.1.2.3. Agaroz Jel Elektroforezinde Kullanılan Kimyasal Maddeler ve Çözeltiler............................................................................................. 32 2.1.3. Primerler..................................................................................................... 33 2.2. Metot................................................................................................................... 33 2.2.1. Morfolojik İncelemeler ............................................................................... 33 2.2.2. Moleküler İncelemeler................................................................................ 35 2.2.2.1. DNA İzolasyonu .................................................................................. 35 2.2.2.2. DNA konsantrasyonlarının belirlenmesi ve muhafaza edilmesi ....... 36 2.2.2.3. Primer seçimi ...................................................................................... 36 2.2.2.4. ISSR reaksiyonları .............................................................................. 36 2.2.2.5. Agaroz jel elektroforezi ve bantların boyanması............................... 37 ix 2.2.2.6. ISSR bantlarının büyüklüğü .............................................................. 37 2.2.2.7. ISSR verilerinin değerlendirilmesi ..................................................... 37 3. BÖLÜM BULGULAR 3.1. Morfolojik Bulgular ........................................................................................... 39 3.2 Moleküler Bulgular ............................................................................................. 48 3.2.1 ISSR verilerine ait Phylogram ağacı........................................................... 50 3.2.2 ISSR verilerine göre hazırlanan Network analizi ...................................... 55 4. BÖLÜM TARTIŞMA VE SONUÇ 4.1. TARTIŞMA ........................................................................................................ 59 4.2 SONUÇ ................................................................................................................ 63 KAYNAKLAR .......................................................................................................... 64 EKLER ...................................................................................................................... 71 ÖZGEÇMİŞ ............................................................................................................ 110 x KISALTMALAR A : Adenin bç : Baz çifti C : Sitozin CTAB : Setil 3-Metil Amonyum Bromid dH₂O : Distile Su DNA : Deoksiribonükleik asit dNTP : Deoksiribonükleik asit trifosfat EDTA : Etilen diamin tetra asetik asit G : Guanin gDNA : Genomik DNA ISSR : Inter-simple Sequence Repeats kb : Kilobaz M : Molar mM : Milimolar MYD :Mehmet Yaşar DADANDI’nın herbaryum koleksiyonu toplayıcı kodu NTSYS : Numerical Taxonomy System ng : Nanogram PVP : Polivinilpirolidon PCR : Polimeraz Zincir Reaksiyonu (Polymerase Chain Reaction) rDNA : Ribozomal DNA TBE : Tris Borat-EDTA u : Ünite V : Volt µl : Mikrolitre UBC : University of British Columbia xi TABLOLAR LİSTESİ Tablo 2.1. Bitki materyallerinin tür adı, toplandığı lokalite ve örnek sayısı ................ 22 Tablo 2.2. DNA izolasyonunda kullanılan kimyasal maddeler ve çözeltiler ............... 31 Tablo 2.3. Polimeraz Zincir Reaksiyonlarında kullanılan kimyasal maddeler ve çözeltiler ................................................................................................... 32 Tablo 2.4. Agaroz jel elektroforezinde kullanılan kimyasal maddeler ve çözeltiler .... 32 Tablo 2.5. ISSR bölgesini amplifiye etmede kullanılan primerler ............................. 33 Tablo 2.6. Morfolojik karakterler ve kodlamaları ...................................................... 34 Tablo 2.7. ISSR-PCR döngüsü .................................................................................. 37 Tablo 3.1. ISSR primerleri ile elde edilen polimorfik bant sayıları ve büyüklükleri .. 49 xii ŞEKİLLER LİSTESİ Şekil 1.1. Kladistik analizinde Aphelandra melezlerinin ebeveyinlerine göre göreceli yerleşimleri. ................................................................................ 12 Şekil 1.2. ISSR-PCR: (TC)n tekrarını hedef alarak zıt yönlerde konumlanmış (TC)n dizileri arasında bulunan inter-SSR bölgesinin (AG)8 primeri ile amplifikasyonunun şematik gösterimi. ...................................................... 20 Şekil 3.1. Phlomis örneklerinde morfolojik verilere ait phylogram ağacı (parça 1) ................................................................................................... 44 Şekil 3.2. Phlomis örneklerinde morfolojik verilere ait phylogram ağacı (parça 2) ................................................................................................... 45 Şekil 3.3 Phlomis örneklerinde morfolojik verilere ait phylogram ağacı (parça 3) ................................................................................................... 46 Şekil 3.4. Phlomis örneklerinin morfolojik verilerinin Manhattan mesafe değerlerine göre oluşturulmuş network analizi 1. Parça ............................. 47 Şekil 3.5 Phlomis örneklerinin morfolojik verilerinin Manhattan mesafe değerlerine göre oluşturulmuş network analizi 2. Parça ............................. 48 Şekil 3.6. ISSR primeri ile ampilifikasyonu yapılan bazı DNA örneklerinin jel görüntüleri ................................................................................................ 49 Şekil 3.7. Phlomis örneklerinde ISSR verileri ile oluşturulan phylogram ağacı parça 1 ...................................................................................................... 52 Şekil 3.8. Phlomis örneklerinde ISSR verileri ile oluşturulan phylogram ağacı parça 2 ...................................................................................................... 53 Şekil 3.9. Phlomis örneklerinde ISSR verileri ile oluşturulan phylogram ağacı parça 3 ...................................................................................................... 54 Şekil 3.10. Phlomis örneklerinde ISSR verilerine ait neighbour- network analizi (1. parça) .................................................................................................. 57 Şekil 3.11. Phlomis örneklerinde ISSR verilerine ait neighbour- network analizi (2. parça) .................................................................................................. 58 1 GİRİŞ Bu çalışmada Phlomis L. cinsine ait türler arasında oluşan doğal melezler ve onların ataları arasındaki filogenetik ilişkiler araştırılmıştır. Melezlerin atalarıyla olan ilişkileri ve türler arası genetik ilişki ISSR markır tekniği ile incelenmiştir. Moleküler verilerin karşılaştırılabilmesi açısından 24 kantitatif karakterde morfolojik ölçümler ve ölçüm sonuçlarına dayalı analizler yapılmıştır. Ayrıca atalarından biri morfolojik verilerle tayin edilemeyen iki farklı P. leucophracta P. H. Davis melezi (MYD 1542 ve 1675) de çalışmaya dahil edilmiştir. Phlomis cinsi Lamiaceae Martinov familyasından otlar, çalılar ve yarı-çalılar içeren yaklaşık 100 türü kapsamaktadır [1]. Türkiye Florası’nda 34 tür, 6 varyete ve 10 doğal melez ile temsil edilmektedir [2]. Dadandı’nın “Türkiye’nin Phlomis L. (Lamiaceae) Cinsi Revizyonu” isimli doktora tezinde bu cins 28 tür, 17 varyete ve 19 doğal melez olarak revize edilmiştir. Bu çalışma ile tür ve varyeteler yeniden düzenlenmiş, 9 yeni doğal melez eklenmiştir [3]. Phlomis cinsi içerisinde yer alan türlerin endemizm oranı %56.41 ve melezlerin endemizm oranı ise %100’dür [4]. Endemizm oranının ve tür sayısının yüksek olması bize cins içerisindeki türleşmenin henüz tamamlanmadığı ve halen devam ettiği kanısını oluşturmaktadır [3]. Phlomis cinsi, Phlomis ve Phlomoides Moench olmak üzere iki ana seksiyona ayrılmıştır [5]. Phlomis seksiyonu 1834 yılında Bentham tarafından yapılan çalışma ile 3 alt seksiyona ayrılmıştır. Bu alt seksiyonlar Oxyphlomis Benth., Dendrophlomis Benth. ve Gymnophlomis Benth. şeklindedir [6]. Oxyphlomis alt seksiyonuna ait türler pembe-mor renkli korollaya sahip, sert ve kalın brakteollüdür. Dendrophlomis alt seksiyonunda türler genellikle sarı nadiren de kahverengi korollalı çok yıllık çalılardır (P. russeliana (Sims.) Lag. ex Benth. hariç). Gymnophlomis alt seksiyonu türleri çok yıllık otsular olup korolla renkleri sarı, brakteolleri incedir [2]. 2 Türkiye'de, Phlomis türleri halk arasında ballık otu, çalba, şalba ve calba olarak isimlendirilmektedir [7]. Kudüs adaçayı, lamba fitili bitkisi ortak isimleri arasındadır [8]. Phlomis ve diğer Lamiaceae familyası türlerinin bal üretiminde önemli bir yeri vardır. Ayrıca Phlomis bitkisinin yaprakları Orta Anadolu, Antalya, Muğla ve Aydın’da çay olarak kullanılmaktadır [9]. Phlomis cinsi türlerinden halk hekimliğinde tonik, gaz giderici, iştah açıcı ve uyarıcı olarak faydalanılmaktadır [10-11]. Familya üyeleri uçucu ve aromatik yağ içermelerinden dolayı farmakoloji ve parfümeri sanayinde önemlidir. Eterik yağ elde edilir, baharat olarak kullanılır ve süs bitkisi olarak yetiştirilir [12]. Phlomis türleri arasında doğal melezleşme oldukça yaygındır. Öyle ki Lamiacea familyası içerisinde en fazla meleze sahip olan cinstir. Tür-türaltı takson endemizmi %58,8’dir ve melez endemizmi %100’dür. Bu durumda, tür-türaltı-melez endemizm oranı %65,3’e varmaktadır [4]. Endemizm oranının yüksek olması Phlomis cinsinin gen merkezlerinden birinin ülkemiz olduğunun göstergesidir [13]. Doğal melezleşme, bitkilerde yaygın olarak oluşur ve bitkilerin türleşmesinde büyük bir role sahiptir [14-15]. Doğada melezleşmeyi kısıtlayıp tür sınırlarının korunmasına yardımcı mekanizmalar mevcuttur. Bu üreme bariyerleri fertilizasyondan önce ya da fertilizayondan sonra etkili olabilir [16]. Bu üreme bariyerlerine rağmen doğal melezleşme bitkilerde türleşmede önemli bir rol oynamaktadır [14-15]. Melezleşme ile bitkiler aleminde kendi içlerinde üreyebilen melezler de ortaya çıkmaktadır. Yeni bir türün doğuşunda türler arası gen akışının büyük rol oynadığı durumlar melez türleşmesi olarak isimlendirilir [17]. Melez ve melez kökenli türlerin bitki türlerinin yaklaşık %25’ini oluşturduğu varsayılmaktadır [18]. Son yıllarda, bitkiler ve onların oluşturduğu toplulukların, ekolojik ve evrimsel süreçlerinin dinamik merkezleri olduklarının kavranmasıyla bitkilerin melezleşme zonları çalışılmıştır [19]. Phlomis L. cinsi türleri arasındaki genetik ilişkiyi ve doğal melezleşmeyi DNA düzeyinde moleküler yöntemlerce inceleyen hem ulusal hem de uluslararası az sayıda çalışma vardır. Phlomis cinsinde türler arası genetik ilişkiyi araştıran, Türkiye dışında yapılan sadece iki uluslarası çalışma mevcuttur. Albaladejo ve arkadaşları 2005 yılında İspanya'da beş farklı Phlomis taksonunda ITS (Internal Transcribed Spacer) markırları ve üç kodlayıcı olmayan plastit DNA bölgesine dayalı olan moleküler çalışmalarında ITS sonuçları ile plastit bölgesine dayalı bulguların çeliştiği belirtilmiş, ayrıca ITS 3 sonuçlarının P. crinita Cav. ve P. lychnitis L.’in ayrılmasında morfolojik ölçüm sonuçlarıyla benzer olduğu belirtilmiştir [20]. Daha kapsamlı olan diğer çalışma ise 2009’da Pan ve ark. tarafından yapılmıştır. Bu çalışmada Çin'de Phlomis'in 16 türü, Lamiophlomis Kudo ‘in 1 türü ve Paraphlomis Prain’in 2 türü üzerinde ITS ve kloroplast DNA (trnL-F ve rpL16) bölgeleri analiz edilmiş ve Lamiophlomis’in Phlomoides seksiyonunda yer aldığı, Phlomis cinsinin Phlomis ve Phlomoides seksiyonlarının monofiletik olduğu ve Paraphlomis’in bağımsız ayrı bir cins olduğu belirtilmiştir [21]. Türkiye'de ise Phlomis türleri arasındaki genetik ilişkiyi ve melezleşmeyi sorgulayan az sayıda çalışma mevcuttur. Yüzbaşıoğlu ve Dadandı 2008 yılında Phlomis’in Dendrophlomis altseksiyonu üyeleri arasında RAPD markır tekniğini kullanarak yaptıkları çalışmada, taksonların çoğunlukla morfolojik sınıflandırmaya benzer bir sıralanma içinde oldukları gözlemlenmiştir [22]. Yüzbaşıoğlu ve ark. P. x termessi P. H. Davis melezinde RAPD primerlerini kullanarak, ataları ile moleküler yönden karşılaştırmış, atalarından aktarılan türlere özgün bantlar ile hem melezliğini hem de ataları ile olan genetik ilişkisini araştırmışlardır. Bu çalışma sonucunda melezin her iki atadan türlere özgün bantlar aldığı ve atalarından birine daha yakın olduğu belirlenmiştir. Ayrıca bu çalışmada, melez bireylerde atalarda olmayan yeni bant oluşumlarının gözlendiği de belirtilmiştir [23-24]. Yüzbaşıoğlu ve ark. bir başka çalışmalarında Phlomis türleri arasındaki genetik ilişkiyi tohumlarında depolanmış olan proteinlerin polimorfizminine dayalı olarak SDS page yöntemi araştırmışlardır. Bu çalışma sonucunda 19’u polimorfik olmak üzere toplamda 21 polipeptit bandı belirlemişlerdir. Bu veriler Phlomis seksiyonunun morfolojik verilere dayalı olarak 3 alt seksiyona (Dendrophlomis, Gymnophlomis, Oxyphlomis) ayrılmış olması durumunu desteklenmektedir. Araştırmada taksonlar arasındaki genetik mesafeler Dendrophlomis seksiyonu için 0.00 ile 0.50 aralığında; Gymnophlomis seksiyonu için 0.00 ile 0.625 aralığında ve Oxyphlomis seksiyonu için 0.00 ile 0.769 aralığında belirlenmiştir [25]. 4 Evren, P. kurdica Rech. f. ve P. oppositiflora Boissieu & Hausskn. türlerinde tür içi genetik çeşitliliği ve bu iki türün melezi olan P. x melitenensis Hub.-Mor.’in melezliğini rastgele çoğaltılmış polimorfik DNA (RAPD) ve basit dizi tekrarları arası bölgelerin çoğaltılması (ISSR) yöntemlerini kullanarak araştırmıştır. Bu çalışma kapsamında P. kurdica ve P. capitata Boiss. türlerinin oluşturduğu P. capitata x kurdica melez örnekleri de moleküler yönden incelenmiştir. Bu çalışma sonucunda elde edilen elektroforez görüntüleri incelendiğinde her iki melezin melezliğini gösteren atalarından melezlere aktarılan türlere özgü bantlar belirlenmiştir. Moleküler verilerde 707’si RAPD’den, 651’i ISSR’dan olmak üzere 1358 bant elde edilmiş, bu bantlardan ikisi hariç geri kalanının polimorfik olduğunu belirtmiştir. RAPD yönteminde en fazla genetik çeşitliliği P. kurdica türünde (H=0.1572; I=0.2646) gözlemişken, ISSR yönteminde ise en fazla genetik çeşitliliği P. capitata (H=0.1403; I=0.2329) türünde gözlemiştir [26]. Sezer, RAPD ve ISSR yöntemlerini kullanarak P. physocalyx Hub.-Mor. ve P. oppositiflora türlerinde tür içi genetik varyasyon düzeyini ve bu iki tür arasındaki melezleşmeyi incelemiştir. Bu çalışmada Nei’nin genetik çeşitlilik indeksi RAPD için 0.14-0.32 aralığında, ISSR için 0.06-0.37 aralığında; Shannon indeksi RAPD için 0.170.49 aralığında, ISSR için 0.06-0.54 aralığında gözlenmiştir [27]. Öztoprak, 15 farklı Phlomis türü arasındaki genetik ilişkiyi ve 12 farklı Phlomis melezinin moleküler yönden atalarına olan yakınlığını kloroplast DNA dizi analizi yöntemiyle incelemiştir. Bu çalışmada kullanılan 43 Phlomis örneğinde nükleotid yüzdeleri %36,84 (A), %35,17 (T/U), %13,28 (C) ve %14,71 (G) olarak belirlenmiştir. Nükleotidlerdeki transisyon/transversiyon dönüşüm hız oranı k1=1.581 (pürinler) ve k2=3.289 (pirimidinler); nükleotidlerdeki toplam transisyon/transversiyon eğilimi ise R=0,958 olarak bulunmuştur [28]. Basit dizi tekrarları arası (ISSR) tekniği PCR’a dayalı bir metottur. Zıt doğrultularda konumlanmış iki özdeş mikrosatellit tekrar bölgesinin arasında amplifiye edilebilir uzaklıkta var olan DNA paçasının amplifikasyonunu kapsar. Bu teknikte primer olarak genellikle 16-25 bç uzunluğundaki mikrosatelitler kullanılır. Tek bir primerli PCR reaksiyonunda, farklı boyutlardaki ISSR dizilerini amplifiye etmek için çoklu genomik lokusları hedef alır [29]. 5 Teknik, AFLP ve mikrosatellit analizlerinin yararlarını, RAPD’in evrenselliğiyle kombine eder. ISSR tekniğinin tekrarlanabilirliği, muhtemelen daha uzun bir primer dizisine (16-25 mer) sahip olması nedeniyle RAPD primerlerine (10-mer) göre daha yüksektir [29]. ISSR basit Mendel kalıtımını izleyen dominant markırlardır. Ancak bazı durumlarda kodominant ayrıldığı gösterilmiştir ve böylece heterozigot ve homozigotu ayırabilir [30]. Mikrosatelitlerdeki evrimsel değişim oranı diğer DNA tiplerine göre oldukça yüksektir, yani bu dizilerde polimorfizmin olabilirliği daha büyüktür. ISSR’lardaki bu değişkenliğin kaynağı kalıp DNA, kullanılan primerlerin doğası ya da tespit yöntemi şıklarından biri ya da birkaçının kombinasyonu olabilir [29]. ISSR tekniği pek çok bitki türündeki filogenetik çalışmalarda; tür içi-türler arası, populasyon içi-populasyonlar arası genetik varyasyon çalışmalarında filogenetik analizlerde yaygın olarak kullanılmaktadır [31-32]. Bu çalışma 36 adet Phlomis melezinin moleküler yönden atalarına olan yakınlığı ve 46 Phlomis taksonu arasındaki genetik ilişkiyi ISSR yöntemiyle incelemek amacıyla yapılmıştır. 6 1. BÖLÜM GENEL BİLGİLER ve LİTERATÜR TARAMASI 1.1. Lamiaceae Familyasının Morfolojik Özellikleri Lamiaceae familyasının üyeleri otlar veya çalılar şeklinde olup, genellikle glandular ve aromatiktirler ve gövdeleri 4 köşeli veya değildir. Yaprakları stipulasız, basit, bazen pinnat, daima opposit, ovat, eliptik ve rotundattır. Temel çiçek durumu brakte veya floral yaprakların koltuğunda taşınan vertisillatlar şeklindedir. Ayrıca vertisillatlar spikamsı baş, rasemus veya kimoz durumlar şeklinde düzenlenmiş olabilir. Çiçekler hermafrodit veya erkek sterildir. Brakteler yapraklara benzer veya belirgin şekilde farklılaşmıştır. Brakteoller mevcut veya eksiktir. Kaliks genellikle 5 loplu, üst lop 3, alt lop 2 dişlidir. Nadiren loplar veya dişler 1-1 veya 1-4 şeklindedir ya da kaliks aktinomoftur. Damarlar 5-20 dir. Korolla gamopetal, zigomorfik ve bilabiat, tüpsü, genellikle üst dudak belirsiz 2 loplu, dik ya da çok az konkav, alt dudak 3 loplu, nadiren üst dudak indirgenmiş ve alt dudak 5 loplu, ya da üstte 1 ve altta 4 loplu, ya da korolla aktinomorfiktir. Stamenler korollaya yapışık, 4 ve didinam ya da 2, üstteki çift genellikle alttaki çiftten daha kısa, anter tekaları 2 ya da 1 gözlü, paralel ya da divergent, nadiren (SalviaL.’de) konnektiflerin uzamasıyla birbirinden ayrılmıştır. Ovaryum üst durumlu, 2 karpelli ve 4 ovüllü, 4 lopludur. Stilus ginobazik, nadiren değil, tepede bifiddir. Meyve 4 (nadiren az) ve kurudur (nadiren etli) [4;33]. 1.2. Phlomis L. Cinsinin Morfolojik Özellikleri Phlomis cinsi türleri çok yıllık otsu veya çalılardır. Yaprakları bölünmemiş, kenarları düz, krenat ya da dentattır. Gövde ve yaprakları yumuşak, dik ve uzunca tüyler (piloz) veya az çok karışmış keçemsi (tomentoz) tüylerle kaplıdır. Salgı tüyleri bulunabilir. Tüyler basit, yıldız şeklinde (stellat) veya ağaçsı (dendroit) olabilir. Vertisillatlar, az veya çok çiçekli, birbirine yakın veya uzaktır. Brakteoller yoktur, varsa az veya çok 7 sayıdadır, subulat-ovat şekilldir. Kaliks tüpsü (tubular) veya darca çan şeklindedir(kampanulat), 5-10 damarlıdır, 5 dişli ve dişler eşit veya değildir. Korolla iki dudaklıdır. Mor, pembe veya sarı renklidir (bazen üst dudak kahverengimsi), korolla tüpü kaliksi aşmaz ve halkalı veya halkasız olabilir. Üst dudak, emerginat; alt dudak patent ve üç lopludur. Stamenler 4 tane, korollanın boyunu aşar veya aşmaz. Anterler çiftler halinde, tekalar divergent ve stilus lopları eşit değildir. Nutletler üç köşelidir, tüylü veya tüysüz olabilir [2]. 1.2.1. Phlomis L. Cinsinin Sistematikteki Yeri Regnum : Plantae Divisio : Spermatophyta Subdivisio : Angiospermae Classis : Magnalopsida Subclass : Asteridae Ordo : Lamiales Familia : Lamiaceae Genus : Phlomis L. [12] 1.2.2. Phlomis L. Cinsi ve Dünya Üzerindeki Yayılışı Phlomis cinsi Lamiaceae familyasından otlar, çalılar ve yarı-çalılar içeren yaklaşık 100 türü kapsamaktadır. Phlomis cinsi, Phlomis ve Phlomoides olmak üzere iki seksiyona ayrılır. Phlomis seksiyonunda korolla iki loptan oluşan kıvrık üst dudağı, ortada büyük bir lop ve yanlarda küçük iki loptan oluşan üç loblu bir alt dudağa sahipken, Phlomoides seksiyonuna ait türlerin korolla yapıları düz üst dudak, eşit loplu trifid alt dudaklıdır. Phlomoides seksiyonu ülkemizde sadece P. tuberosa L. ile temsil edilmektedir. Phlomis seksiyonu ise Gymnophlomis, Dendrophlomis ve Oxyphlomis olmak üzere 3 altseksiyona ayrılır. Dendrophlomis altseksiyonunda brakteoller linear-subulattan 8 lanseolat ve ovata kadar olan şekillerde ve çok sayıdadır. Oxyphlomis seksiyonunda çoğu tür sert, kaliksle eşit (bazen daha uzun) linear-subulat, çok sayıda brakteollüdür. Gymnophlomis alt seksiyonunda brakteoller zayıf, az ya da çok sayıda nadiren yok, linear-subulat, küçük (2-10 mm) tabanda bağımsızdır [1]. Phlomis cinsine ait türler Akdeniz bölgesinden orta Asya ve Çin'e kadar yayılır. Phlomoides seksiyonu türleri daha çok orta Asya ve Çin’de yayılış gösterirken, Phlomis seksiyonu türleri ağırlıklı olarak Akdeniz bölgesinde yayılış gösterir. Ülkemizde Phlomis seksiyonundan yaklaşık 34 tür ve bu türlerde %57 endemizm oranı görülmektedir. Bu da Türkiye’yi Phlomis seksiyonun Akdeniz bölgesindeki çeşitlenme merkezlerinden biri olarak göz önüne getirmiştir. Özellikle Türkiye’nin güney ve doğu bölümleri ile İran’ın kuzeybatı bölümünün bu seksiyonun orjin merkezleri olduğu ileri sürülmüştür. İran için Phlomis seksiyonuna ait tür sayısı 18 ve bu türlerde görülen endemizm oranı %33’tür [13]. Türkiye Florasında Phlomis cinsi 33 tür, 6 varyete ve 12 doğal melez ile temsil edilmektedir. Bu türlerin 4 tanesi Oxyphlomis, 13 tanesi Gymnophlomis ve 15 tanesi Dendrophlomis altseksiyonlarına yerleştirilmiştir [34]. Dendrophlomis altseksiyonuna ait türler arasından P. amanica Vierh., P. bourgaei Boiss., P. chimerae Boissieu, P. grandiflora var. fimbrilligera (Hub.-Mor.) Hub.-Mor., P. leucophracta, P. longifolia var. bailanica (Vierh.) Hub.-Mor., P. lycia D. Don., P. monocephala P. H. Davis ve P. russeliana’yı kapsayan 9 tür Türkiye Florası’na göre endemiktir [2]. P. amanica nesli tehlike altında, P. grandiflora var. fimbrilligera zarar görebilir olarak kabul edilmiş, diğerleri düşük risk altında bulunmuştur [35]. P. chimerae ve P. amanica sırasıyla sadece Çıralı (Antalya) ve Arsuz (Hatay) civarında yetişen yerel endemikler olarak bilinir [3]. 1.2.3. Phlomis L. Cinsi Üzerine Yapılan Çalışmalar Phlomis taksonları arasındaki ilişkileri aydınlatmak için morfolojik, anatomik, palinolojik ve sitolojik özellikleri kullanan çok sayıda çalışma mevcuttur [1-3;20-28;3638]. Ancak türler arasındaki genetik ilişkiyi ve doğal melezleşmeyi moleküler yöntemlerce inceleyen hem ulusal hem de uluslararası az sayıda çalışma vardır. 9 Phlomis cinsinde türler arası genetik ilişkiyi araştıran, Türkiye dışında yapılan sadece iki uluslarası çalışma mevcuttur. Albaladejo ve arkadaşları 2005 yılında İspanya'da beş farklı Phlomis taksonunda ITS (Internal Transcribed Spacer) markırları ve üç kodlayıcı olmayan plastit DNA bölgesine dayalı olan moleküler çalışmalarında ITS sonuçları ile plastit bölgesine dayalı bulguların çeliştiği belirtilmiş, ayrıca ITS sonuçlarının P. crinita ve P. lychnitis’in ayrılmasında morfolojik ölçüm sonuçlarıyla benzer olduğu belirtilmiştir [20]. Daha kapsamlı olan diğer çalışma ise 2009’da Pan ve arkadaşları tarafından yapılmıştır. Bu çalışmada Çin'de Phlomis'in 16 türü, Lamiophlomis'in 1 türü ve Paraphlomis’in 2 türü üzerinde ITS ve kloroplast DNA (trnL-F ve rpL16) bölgeleri analiz edilmiş ve Lamiophlomis’in Phlomoides seksiyonunda yer aldığı, Phlomis cinsinin Phlomis ve Phlomoides seksiyonlarının monofiletik olduğu ve Paraphlomis’in bağımsız ayrı bir cins olduğu belirtilmiştir [21]. Türkiye'de ise Phlomis türleri arasındaki genetik ilişkiyi ve melezleşmeyi sorgulayan az sayıda çalışma mevcuttur. Yüzbaşıoğlu ve Dadandı 2008 yılında Phlomis’in Dendrophlomis altseksiyonu üyeleri arasında RAPD markır tekniğini kullanarak yaptıkları çalışmada, taksonların çoğunlukla morfolojik sınıflandırmaya benzer bir sıralanma içinde oldukları gözlemlenmiştir [22]. Yüzbaşıoğlu ve ark. P. x termessi melezinde RAPD primerlerini kullanarak, ataları ile moleküler yönden karşılaştırmış, atalarından aktarılan türlere özgün bantlar ile hem melezliğini hem de ataları ile olan genetik ilişkisini araştırmışlardır. Bu çalışma sonucunda melezin her iki atasından türlere özgün bantlar aldığını ve atalarından birine daha yakın olduğu olduğu belirlenmiştir. Ayrıca bu çalışmada, melez bireylerde atalarda olmayan yeni bant oluşumlarının gözlendiği de belirtilmiştir [23-24]. Yüzbaşıoğlu ve ark. bir başka çalışmalarında Phlomis türleri arasındaki genetik ilişkiyi tohumlarında depolanmış olan proteinlerin polimorfizminine dayalı olarak SDS page yöntemi araştırmışlardır. Bu çalışma sonucunda 19’u polimorfik olmak üzere toplamda 21 polipeptit bandı belirlemişlerdir. Bu veriler Phlomis seksiyonunun morfolojik verilere dayalı olarak 3 alt seksiyona (Dendrophlomis, Gymnophlomis, Oxyphlomis) ayrılmış olması durumunu desteklenmektedir. Araştırmada taksonlar arasındaki genetik mesafeler Dendrophlomis seksiyonu için 0.00 ile 0.50 aralığında; Gymnophlomis 10 seksiyonu için 0.00 ile 0.625 aralığında ve Oxyphlomis seksiyonu için 0.00 ile 0.769 aralığında belirlenmiştir [25]. Evren, P. kurdica ve P. oppositiflora türlerinde tür içi genetik çeşitliliği ve bu iki türün melezi olan P. x melitenensis’in melezliğini rastgele çoğaltılmış polimorfik DNA (RAPD) ve basit dizi tekrarları arası bölgelerin çoğaltılması (ISSR) yöntemlerini kullanarak araştırmıştır. Bu çalışma kapsamında P. kurdica ve P. capitata türlerinin oluşturduğu P. capitata x kurdica melez örnekleri de moleküler yönden incelenmiştir. Bu çalışma sonucunda elde edilen elektroforez görüntüleri incelendiğinde her iki melezin melezliğini gösteren atalarından melezlere aktarılan türlere özgü bantlar belirlenmiştir. Moleküler verilerde 707’si RAPD’den, 651’i ISSR’dan olmak üzere 1358 bant elde edilmiş, bu bantlardan ikisi hariç geri kalanının polimorfik olduğunu belirtmiştir. RAPD yönteminde en fazla genetik çeşitliliği P. kurdica türünde (H=0.1572; I=0.2646) gözlemişken, ISSR yönteminde ise en fazla genetik çeşitliliği P. capitata (H=0.1403; I=0.2329) türünde gözlemiştir [26]. Sezer, RAPD ve ISSR yöntemlerini kullanarak P. physocalyx ve P. oppositiflora türlerinde tür içi genetik varyasyon düzeyini ve bu iki tür arasındaki melezleşmeyi ile incelemiştir. Bu çalışmada Nei’nin genetik çeşitlilik indeksi RAPD için 0.14-0.32 aralığında, ISSR için 0.06-0.37 ralığında; Shannon indeksi RAPD için 0.17-0.49 aralığında, ISSR için 0.06-0.54 aralığında gözlenmiştir [27]. Öztoprak, 15 farklı Phlomis türü arasındaki genetik ilişkiyi ve 12 farklı Phlomis melezinin moleküler yönden atalarına olan yakınlığını kloroplast DNA dizi analizi yöntemiyle incelemiştir. Bu çalışmada kullanılan 43 Phlomis örneğinde nükleotid yüzdeleri %36,84 (A), %35,17 (T/U), %13,28 (C) ve %14,71 (G) olarak belirlenmiştir. Nükleotidlerdeki transisyon/transversiyon dönüşüm hız oranı k1=1.581 (pürinler) ve k2=3.289 (pirimidinler); nükleotidlerdeki toplam transisyon/transversiyon eğilimi ise R=0,958 olarak bulunmuştur [28]. 1.3. Bitkiler Aleminde Melezleşme Melez kavramı farklı türlerlerden bireylerin arasındaki çarpraz döllenme sonucu oluşmuş organizmalar olarak açıklanabilir. Evrimsel biyologlara göre melezleşmenin 11 birçok tanımı vardır ancak melezleşme genel anlamda, bir ya da daha fazla kalıtsal karakter bazında birbirinden ayrılabilen iki populasyon ya da populasyon gruplarından bireylerin birleşmesi olarak tanımlanır [15]. Bitkiler aleminde türler arası birleşme sonucu oluşan melezler kısır olabilirler. Nadiren üretilebilen verimli polenler olsa da tohum oluşturulamaz ve bu nedenle üreyemezler [37]. Melezleşme kontrollü koşullar altında yapay olarak oluşturulabilir. Ayrıca doğal şartlar altında kendiliğinden meydana gelebilir. Doğada melezleşmeyi kısıtlayıp tür sınırlarının korunmasına yardımcı mekanizmalar mevcuttur. Bu üreme bariyerleri fertilizasyondan önce ya da fertilizayondan sonra etkili olabilir [16]. Bu üreme bariyerlerine rağmen doğal melezleşme bitkilerde türleşmede önemli bir rol oynamaktadır [14-15]. Melezleşme ile bitkiler aleminde kendi içlerinde üreyebilen melezler de ortaya çıkmaktadır. Yeni bir türün doğuşunda türler arası gen akışının büyük rol oynadığı durumlar melez türleşmesi olarak isimlendirilir [17]. Melez ve melez kökenli türlerin bitki türlerinin yaklaşık %25’ini oluşturduğu varsayılmaktadır [18]. Son yıllarda, bitkiler ve onların oluşturduğu toplulukların, ekolojik ve evrimsel süreçlerinin dinamik merkezleri olduklarının kavranmasıyla bitkilerin melezleşme zonları çalışılmıştır [19]. McDade Aphelandra L. cinsinin 12 türünde ve bu türler arasında kontrollü melezlemelerle oluşturduğu 17 melezinde 50 morfolojik karaktere dayalı olarak oluşturduğu kladistik analizde melez bireylerin ebeveyinlerine göre kladistikteki yerleşimlerini 4 kategoride toplamıştır: (1) Melez ebeveyinleri arasında yerleşmiştir. Bu yerleşme ebeveyinlerden biriyle kardeş (sister) takson olacak şekilde (Şekil 1a, 1b) veya yine ebeveyinlerden biriyle fakat kardeş taksondan farklı bir şekilde yerleşmiştir (Şekil 1c, 1d). (2) Melez en son türevlenen ataya tabandan bağlanmıştır (Şekil 1e). (3) Melez en son türevlenen ataya kladistik olarak yakın bir şekilde yerleşmiştir (Şekil 1f, 1g). (4) Melez diğer melezler/taksonlarla bir arada gruplanmıştır (Şekil 1h, 1I, 1j) [39]. PCR’a dayalı moleküler markırlarla genetik varyasyon çalışmalarının elektroforez görüntüleri incelendiğinde atalarında gözlenebilen türe özgü bantların melezlerde de varlığı saptanmıştır. Bu bant atalardan yalnız birinde var olabilirken her ikisinde de mevcut olabilir. Bunun yanı sıra melez bireylerde krossing-overdan kaynaklı genetik rekombinasyonlar dolayısıyla açığa çıkan meleze özgü yeni bantların varlığı da gözlenmiştir. [23]. 12 Şekil 1.1. a-j. Kladistik analizinde Aphelandra melezlerinin ebeveyinlerine göre göreceli yerleşimleri. (CA) Aphelandra campanensis; (DA) A. darienensis.; (GO) A. golfodulcensis; (GR) A. gracilis; (HA) A. hartwegiana; (LE)A. leonardii; (LI) A. lingua-bovis; (PA) A. panamensis; (SC) A. scabra; (SI) A. sinclairiana; (ST) A. storkii; (TE) A. terryae. Melezler: GO X LE, GO X SC, GO X SI, LE X CA, LE X GO, LE X SI, PA X GO, PA X LE, PA X SC, PA X SI, SC X GO, SC X PA, SC X SI, SC X ST, SI X GR, SI X SC, SI X TE [40] 13 1.4. Moleküler Sistematik 17. yüzyılda Linnaeus tarafından yapılan hiyerarşik tabanlı sistem, dünyadaki biyolojik çeşitliliği anlamak ve değerlendirmek için yapılan sınıflandırmalara temel oluşturmuştur. Linnaeus’tan sonraki çalışmalarda ise filogenetik akrabalıkların öneminin ön plana çıkmasıyla filogenetik sistematik yöntemi sistematikte yer almaya başlamıştır [40]. 1960’larda DNA’nın keşfiyle bu alandaki çalışmalar hız kazanmıştır [41]. 1980’lerin ortalarından itibaren PCR teknolojisinin yardımıyla genomun tanımlanması ve seçilmesi hızla ilerlemiştir. Sadece küçük miktarlarda DNA’ya ihtiyaç duyan hızlı ve tekrarlanabilir birçok markır protokolü geliştirilmiştir [42]. Kalıtımları morfolojik, biyokimyasal, protein ve DNA düzeyinde izleyebilen karakterlere “markır” denir [43]. Her markır tekniğinin kendine özgü avantajları ve dezavantajları vardır. Moleküler sistematik alanındaki ilerlemeler morfoloji temelli sistematik yaklaşımını tamamen devre dışı bırakmasa da filogenetik çalışmalarda moleküler düzeyde bir yaklaşımın daha güçlü sonuçlar elde edeceğini ortaya koymuştur [41]. Markırlar; morfolojik markırlar, biyokimyasal markırlar ve moleküler markırlar olmak üzere üç grupta toplanır. 1.4.1. Morfolojik Markırlar Populasyonların genetik yapısının belirlenmesi için yapılan ilk çalışmalar morfolojik özelliklere dayanmaktadır. Tek lokus ile idare edilen morfolojik özellikler, değişik çevre koşullarında ifade edilebildiği sürece genetik markır olarak kullanılabilir. Ancak genetik çeşitliliğin tamamı morfolojiye yansımaz [44]. Bu markırların avantajlı yanları analizlerinin kolay olması, ucuz olmaları ve haritalamanın kolayca yapılabiliyor olmasıdır. Sayılarının az olması, çevresel faktörlerden etkilenmeleri ise dezavatajlarından bazılarıdır. Morfolojik markırlar genellikle dominant özelliktedir ve bu nedenle dominant (AA veya Aa) ve resesif (aa) fenotipleri belirleyebilirken, heterozigot (Aa) fenotipleri dominant fenotiplerden ayırt edemezler [43;45]. Ayrıca morfolojik karakterlerdeki her varyasyon genetik tabanlı değildir [20]. 14 1.4.2. Biyokimyasal Markırlar Protein markırları, DNA teknolojisinin henüz gelişmediği dönemlerde önemli ilerlemeler sağlamıştır [44]. Bu yöntem, proteinlerin elektroforetik olarak ayrılmasına ve bu proteinlerin substratları ile enzimatik olarak boyanmasını temel alır [46]. Kodominant özellikte olduklarından saptanan farklılıklar farklı genotiplerdeki çeşitliliğe işaret eder [43;47]. Biyokimyasal markırlar depo ve enzim proteinleri olmak üzere iki ana gruba ayrılırlar. Depo proteinleri bir jelde hareket ettirilip belirli boyalarla boyandıklarında farklı genotiplerde ortaya çıkan farklı bantlar genetik markır olarak kullanılabilir. Bu markırların avantajı analizlerinin çabuk, tekrarlanılabilir ve güvenilir olmasıdır [43]. Enzim markırları izoenzimler ve alloenzimler olmak üzere iki gruba ayrılır. Belli bir enzimin katalitik aktivitesi aynı, fakat elektriksel alanda göçü, doku dağılımı, inhibitör ve aktivatörlere yanıtları farklı olan formlarına o enzimin “izoenzimleri” denir. İzoenzimler, amino asit dizinleri farklı olduğu halde aynı tepkimeyi katalize eden enzimlerdir; farklı dokularda veya farklı hücre içi kompartımanlarda farklı rollere sahiptirler. Eğer bir reaksiyon hücrenin hem sitzolünde hem mitokondrisinde gerçekleşiyorsa, genellikle farklı izoenzimler tarafından katalize edilir [43]. Alloenzimler ise aynı lokustaki farklı alleler tarafından kodlanan bir enzimin farklı moleküler formlarıdır [48]. İzoenezimlerin metobolizmada üstlendikleri rollerin iyi bilinmesi, kullanılan yöntemlerin ucuz ve hızlı olması, izoenzim markırlarını avantajlı hale getirmektedir. Ancak az sayıda lokus olması, bazı izoenzimlerin belirli dokulara spesifik olması veya belli dönemlerde ifade edilebiliyor olması ve translasyon sonrası modifikasyonlara uğraması bu markırların temel dezavantajlarıdır [47;49]. 1.4.3. Moleküler Markırlar Moleküler markırlar, genom içinde bir DNA parçasının eklenmeler, silinmeler, yer değiştirmeler, duplikasyonlar gibi olaylar sonucu meydana gelen farklılıklarını ortaya koyarlar. DNA temelli moleküler markırlar taksonomi, fizyoloji, embriyoloji, genetik mühendisliği vb. alanlarda kullanılan çok yönlü araçlardır [55]. Kloroplast DNA’sı 15 (cpDNA), mitokondriyel DNA (mtDNA) ve çekirdek DNA’sı (nDNA) genomları DNA markır sisteminde kullanılabilir [50]. Bu markırlar; (1) Fonksiyonu bilinen genlere veya genomun herhangi bir bölgesine özgü proplarla Southern Blot hibridizasyonuna ve kesim enzimlerine dayalı olan RFLP markırları, (2) RAPD metodu gibi PCR temelli metotlar, (3) AFLP gibi hem kesim enzimlerine hem de PCR metoduna dayalı olan metotlar, (4) DNA dizi analizli gerektirmekte olan ISSR, SSR ve SCAR gibi metotlar olmak üzere dört grupta sınıflandırılmaktadır [51]. İdeal bir moleküler markır tekniğinin; polimorfik olması ve genom boyunca dağılması, genetik farklılıkların ortaya çıkarılmasında yeterli olması, çok sayıda, bağımsız ve güvenilir markırlar üretmesi, basit, hızlı ve ucuz olması, az miktar DNA veya doku ihtiyacı gerektirmesi, farklı fenotiplerle bağlantı oluşturması gibi özelliklere sahip olması avantajdır ama hiçbir markır tekniği bu avantajların tümüne birden sahip değildir [52-53]. 1.4.3.1. Restriksiyon Parça Uzunluk Polimorfizmi - Restriction Fragment Lenght Polymorphism (RFLP) Hibridizasyon temelli bir markır tekniğidir. 1983 yılında geliştirilmiştir ve geliştirilen ilk DNA markırıdır. DNA’nın restriksiyon endonükleaz enzimi ile kesimiyle oluşturulan parçalarının uzunluk farkının belirlenmesi prensibini esas alır. Kesim bölgesi ortadan kaldıran tek baz değişiklikleri en yaygın olarak gözlenen mutasyondur. Bu yöntemde populasyonlar arasında veya içindeki kesim bölgesinde meydana gelebilecek mutasyonlar ya da DNA eşleşmesi sonunda ortaya çıkan hatalar RFLP analizi ile tanımlanabilir [54]. RFLP tekniğinin avantajları yüksek oranda tekrarlanabilir olması, kodominant katılması ve heterozigot bireylerin tanınmasının sağlamasıdır. Ancak, DNA amplifikasyonuna dayalı moleküler tekniklere göre pahalı olması, radyoaktif madde kullanılması, zaman alıcı ve fazla miktarda DNA gerektirmesi ve uygulanmasının zor olması gibi dezavantajları vardır [55]. 1.4.3.2. Çoğaltılmış Parça Uzunluk Polimofizmi - Amplified Fragment Lenght Polymorphism (AFLP) AFLP markır tekniği, kesim enzimleri ile oluşturulmuş DNA parçalarının seçici amplifikasyonuna dayanmaktadır ve PCR ile RFLP tekniğinin birleştirilmesiyle ortaya 16 çıkmıştır. Kullanılan primerler hem diziye özgü hem de rastgele olabilir [56]. Bu yöntem genomun bütününde DNA polimorfizmini tespit etmek için kullanılır [57]. AFLP tekniğinin avantajları; polimorfizm oranının yüksek olması, genomik DNA’nın bilinmesine gerek olmaması, RFLP’ye göre hızlı olması, minimum primer testi ile çok sayıda markır üretmesi, parmak izi analizleri için uygun olması, sayıları RAPD ve RFLP’den fazla olması ve QTL analizleri için uygun olmasıdır. Tekniğin dezavantajları ise; saf ve yüksek moleküler ağırlıkta DNA’ya gereksininim duyulması, pahalı bir teknik olması, genellikle dominant markırlar vermesi ve farklı genetik haritalar arasında transferinin güç olmasıdır [58]. 1.4.3.3. Rastgele Çoğaltılmış Polimorfik DNA - Randomly Amplified Polymorphic DNA(s) (RAPD) RAPD tekniği PCR’a dayalı bir yöntemdir. Bu yöntemde her tür organizmanın DNA’sı kullanılabilir. Teknik 6-10 bazlık tek bir primer ile genomik DNA’da ilgili bölgeler çoğaltılmasını esas alır. Bu primerler nükleotid dizisi rastgele belirlenen kısa zincirli oligonükleotidlerden oluşur ve genom üzerinde rastgele bölgelerin DNA amplifikasyonu yapılır. Belirli bir DNA sırasına ait bilgi gerektirmemektedir. RAPD ürünleri genellikle agaroz jelde yürütülür. Jelin boyanmasında ethidyum bromür veya gümüş nitrat kullanılır. Amplifiye edilmiş bölgelerdeki uzunluk farkı bantları birbirinden ayırt eder. RAPD tekniği; hızlı sonuç vermesi, polimorfizm oranının yüksek olması, maliyetinin düşük olması, az işgücü gerektirmesi, az miktarda DNA’ya gereksinim duyulması, amplifikasyon için ön dizi bilgisine ve bantların görüntülenmesinde radyoaktif izotoplara gereksinim duyulmaması açısından avantajlıdır. Tekniğin dezavantajları ise tekrarlanabilirliğinin düşük olması ve dominant kalıtım göstermesidir [59]. 1.4.3.4. DNA Dizisi Belirlenerek Çoğaltılmış Polimorfik Bölgeler - Sequence Characterized Amplified Regions (SCAR) SCAR yöntemi dizi analizi ile sekansı belirlenmiş RAPD ürünlerinin ilk 10 bazının arkasına, dizi bilgisi dikkate alınarak iç dizilerden oluşturulmuş 14 bazın eklenmesiyle tasarlanan 24 bazlık bir primerle DNA’nın amplifikasyonuna dayanır. Genetik açıdan tek bir lokus olarak tanımlanan genomik DNA fragmentlerini karakterize eder. 17 Genellikle dominant özellik gösterir ancak SCAR markırları kesim enzimleri ile kesilerek kodominant markırlara dönüştürülebilmektedir. Tekrarlanılabilirliği, RAPD ve ISSR markırlarına göre çok daha yüksektir. Çok düşük miktarda da olsa dizi bilgisi gerektirir [60]. 1.4.3.5. İçsel Transkribe Edilen Bölge - Internal Transcribed Spacer (ITS) Bu teknik ribozomal DNA üzerindeki ITS bölgelerinin çoğaltılması esasına dayanır. rDNA lokusu 5’ den 3’ ne doğru okunduğunda 5’ETS, 18s rRNA, ITS1, 5.8S rRNA, ITS2, 26S rRNA ve son olarak da 3’ ETS kısımlarından oluşmaktadır. Amplifikasyon için iki farklı primer kullanılır. Monomorfik bant verir ve dizi analizi gerektirir. Bu bölgenin uzunluğu kapalı tohumlu bitkilerde 500–700 bç arasında değişiklik gösterirken açık tohumlu bitkilerde bu bölgenin uzunluğu 1500 ile 3500 baz çiftine kadar çıkabilir. [61-62]. ITS dizileri canlılar arasındaki filogenetik ilişkilerin belirlenmesi, genetik varyasyon çalışmaları ve melezlerin atalarıyla moleküler açıdan karşılaştırılmasında yaygın olarak kullanılır. Güvenilirliği oldukça yüksektir [63]. 1.4.3.6. Kloroplast DNA (cpDNA) Kloroplast DNA’sı çoğunlukla tek bir ebeveynden kalıtılır. Kloroplast DNA’sı üzerinde bulunan trnT-trnF, atpB, psbA, trnH gibi bölgeler ve rpL16 intronu filogenetik çalışmalarda yaygın olarak kullanılmaktadır. Çoğaltılan bölgelerde dizi analizi yapılır ve dizilerdeki farklılıklar filogenetik ilişkilerin belirlenmesinde kullanılır [32]. Bu teknik kloroplast DNA’sındaki hedef bölgenin bir çift primerle amplifikasyonuna dayanır. PCR ürünleri agaroz jelde yürütülür ve ethidyum bromür ile boyanarak görüntülenir. Monomorfik bant verir. Dizi bilgisi gerektirmesi nedeniyle maliyeti ve güvenilirliği yüksektir [20]. 1.4.3.7. Basit Dizi Tekrarları (Mikrosatallitler) - Simple Sequence Repeats (SSR) Bu yöntem STR (short tandem repeats) olarak da adlandırılır. Mikrosatellitler yüksek organizmalarda bulunan, 2-6 uzunluğundaki (AT), (GT), (ATT), (CTT) veya (GATA) gibi nükleotidlerin n sayıda tekrarlarından oluşurlar. Mikrosatelitler yüksek organzmalara ait kromozomlarda daha bol ve gelişigüzel dağılım gösterirler. Görevleri tam olarak bilinmemekle birlikte düzenleyici rollere sahip olduğu düşünülmektedir [64]. 18 Mikrosatelitleri çevreleyen DNA dizileri genellikle aynı türün bireyleri arasında korunmuş olmaları nedeniyle farklı genotipte çakışan mikrosetelitlerin PCR ile çoğaltılarak seçimine izin vermektedir. Ardışık tekrarların sayısındaki farklılık PCR sonucu farklı uzunlukta parçaların amplifikasyonu ile sonuçlanır. Bu tekrarlar çok yakın tür ve çeşitler arasında dahi tekrarlanan ünitelerin sayısında değişikliğe neden olan mutasyonlar sebiyle oldukça polimorfiktir [65]. Bu sayede ökaryotik genomda polimorfizmin mükemmel kaynakları olup genetik çalışmalarında oldukça etkili olmuştur [47]. Mikrosatallit DNA markırları, popülasyon düzeyindeki, araştırmalarda son derece kullanışlıdır. Bu yöntemin avantajları; polimorfizm oranının ve sonuçların tekrarlanabilirliğinin çok yüksek olması ve heterozigot ve homozigotu ayırt edebilmesidir (kodominant markır). Bu yöntemin dezavantajı ise çoğaltılmak istenen mikrosatelit lokusunun etrafındaki dizilerin bilinmesi gerektirmesidir [66]. 1.4.3.8. Basit Dizi Tekrarları Arası - Inter Simple Sequence Repeats (ISSR) Basit dizi tekrarları arası (ISSR) tekniği PCR’a dayalı bir metottur. Zıt doğrultularda konumlanmış iki özdeş mikrosatellit tekrar bölgesinin arasında amplifiye edilebilir uzaklıkta var olan DNA paçasının amplifikasyonunu kapsar. Bu teknikte primer olarak genellikle 16-25 bç uzunluğundaki mikrosatelitler kullanılır. Tek bir primerli PCR reaksiyonunda, farklı boyutlardaki ISSR dizilerini amplifiye etmek için çoklu genomik lokusları hedef alır. Primer olarak kullanılan mikrosatelitler di-nükleotid, tri-nükleotid, tetra-nükleotid ya da penta-nükleotid olabilir [29]. Kullanılan primerler uçlarından 2-4 baz uzatılabilir. Bu uzatılan uçlar 3’ uçlarıdır [67]. Bu uzantılar içindeki kısımlarda seçici bazlar bulunması sayesinde çok sayıda lokus aynı anda izlenebilir [68]. Bu seçici bazlar içerisinde “R” ve “Y” ile gösterilen gruplar bulunur. Burada “R” DNA’da bulunan pürinlerden adenin veya guaninden herhangi birinin; “Y” ise DNA’da bulunan pirimidinlerden sitozin veya timinden herhangi birinin geçebileceğini gösterir [67]. Teknik, AFLP ve mikrosatellit analizlerinin yararlarını, RAPD’in evrenselliğiyle kombine eder. ISSR tekniğinin tekrarlanabilirliği, muhtemelen daha uzun bir primer dizisine (16-25 mer) sahip olması nedeniyle RAPD primerlerine (10-mer) göre daha yüksektir. Bu da daha yüksek yapışma sıcaklığını (45-60 C) müteakiben daha yüksek 19 seçicilikte yapışmaya izin verir. Yapışma sıcaklığı kullanılan primerin GC içeriğine bağlıdır ve genellikle 45 - 60 C arasında değişir [29]. ISSR basit Mendel kalıtımını izleyen dominant markırlardır. Ancak bazı durumlarda kodominant ayrıldığı gösterilmiştir ve böylece heterozigot ve homozigotu ayırabilir [30]. Mikrosatelitlerdeki evrimsel değişim oranı diğer DNA tiplerine göre oldukça yüksektir, yani bu dizilerde polimorfizmin olabilirliği daha fazladır. ISSR’lardaki bu değişkenliğin kaynağı kalıp DNA, kullanılan primerlerin doğası ya da tespit yöntemi şıklarından biri ya da birkaçının kombinasyonu olabilir [29]. ISSR ürünlerinin görüntülenmesinde radyoaktiviteyle (PCR reaksiyonunda etiketlenmiş nükleotid kullanılarak) kombine edilen poliakrilamid jel elektroforezi (PAGE) en yüksek hassasiyeti göstermiştir. Bu yöntemi mütakiben gümüş ile boyanmış poliakrilamid jel elektroforezi ve bundan sonra agaroz-etidyum bromid sistemiyle tespit metodu gelmiştir. Agarozla karşılaştırıldığında poliakrilamid jelde primer başına çözülen bant sayısı belirgin derecede yüksektir [69]. ISSR-PCR basit, hızlı ve etkili bir tekniktir. Tekrarlanabilirliği yüksektir. Radyoaktivite kullanımı esansiyel değildir. Primerlerin ön dizi bilgisi gerektirmemesi, optimizasyonunun kolay olması ve uzun olması (16-25 bç) nedeniyle de primerin doğru bölgeye yapışma ihtimalinin yüksek olması avantajdır. Amplifiye edilmiş ürünler genelde 200-2000 bç uzunlukta olur ve hem poliakrilamid hem de agaroz jel elektroforezi ile tespit edilebilir [29]. ISSR tekniği pek çok bitki türündeki filogenetik çalışmalarda; tür içi-türler arası, populasyon içi-populasyonlar arası genetik varyasyon çalışmalarında filogenetik analizlerde yaygın olarak kullanılmaktadır [31-32]. 20 Şekil 1.2. ISSR-PCR: (TC)n tekrarını hedef alarak zıt yönlerde konumlanmış (TC)n dizileri arasında bulunan inter-SSR bölgesinin (AG)₈ primeri ile amplifikasyonunun şematik gösterimi. Uzatılmamış primer (a) kalıp DNA üzerinde tekrar dizisindeki herhangi bir yere bağlanabilir bu da kaymaya ve smear oluşumuna neden olur. 3’ ucundan iki nükleotit (NN) uzatılan primer (b) kalıp DNA üzerinde spesifik bölgelere yapışır ve temiz bantlar üretir. 5’ ucundan 2 nükleotid uzatılmış primer (c) spesifik bölgelere yapışır ve daha büyük bantlar üretmesini sağlayan bölgeyi çoğaltır [29]. 21 2. BÖLÜM MATERYAL VE METOD 2.1. Materyal 2.1.1. Bitki Materyali Bu çalışmada, Erciyes Üniversitesi Biyoloji Bölümü herbaryumunda korunan bitki örnekleri kullanılmıştır. Bu bitki materyalleri başta Mehmet Yaşar Dadandı olmak üzere farklı araştırmacılar tarafından çeşitli lokalitelerden toplanmış ve morfolojik özelliklerine göre teşhis edilmiş ve ait oldukları taksonlar belirlenerek etiketlenmiştir. Araştırmada kullanılan Phlomis melezleri aşağıda verilmiştir. Bu çalışmada kullanılan bitki materyalleri, örnek numaraları ve toplanma tarihleri ile Tablo 2.1 de verilmiştir. Phlomis x alanyensis Hub.-Mor. (P. leucophrachta x lunariifolia Sm.), P. x bornmuelleri Rech. f. (P. armeniaca Wild. x nissolii L.), P. x cilicica Hub.-Mor. (P. lunariifolia x monocephala), P. x ekimii Dadandı & H. Duman ( P. brugueiri Desf. x P. capitata), P. x kalanensis Hub.-Mor. (P. linearis Boissieu & Balansa x oppositiflora), P. x ketenoglui Dadandi & H. Duman (P. capitata x nissolii), P. x melitenensis (P. kurdica x oppositiflora), P. x mobullensis Hub.-Mor. (P. bourgaei x grandiflora H. S. Thomps), P x muglensis Hub.-Mor. (P. grandiflora x P. lycia), P. x ozhatayii Dadandi & H. Duman. (P. kurdica x capitata), P. x rechingeri Hub.-Mor. (P. armeniaca x carica Rech. f.), P. x semiorbata Rech. f. (P. capitata x linearis), P. x termessi P. H. Davis (P. lycia x bourgaei), P. x tunceliensis Hub.-Mor. (P. kurdica x linearis), P. x turcica Dadandi & H. Duman (P. armeniaca x linearis),P. x vuralii Dadandi (P. bourgaei x chimera), P. physocalyx x oppositiflora, P. capitata x brunneogaleata Hub.-Mor., P. brugueiri x brunneogaleata, P. pungens Willd. x nissolii. 22 Tablo 2.1. Bitki materyallerinin tür adı, toplandığı lokalite ve örnek sayısı Herbaryum Numarası Tarih Tür/melez adı Lokalite 1 P. bourgaei 21.06.1997 2 P. lycia 3 P. leucophrocta Antalya: Termessos Milli Parkı 1002 MYD 500 m Antalya: Termessos Milli Parkı 1006 MYD 500 m Antalya: Manavgat-Alanya arası 1017-2 MYD Kestel’in 12 km kuzeyi yol kenarı 700 m 4 P. tuberosa Sivas: İmranlı-Zara 10. çeşmenin üzeri 1350 m km 1019-4 MYD 14.07.1997 5 P. tuberosa Sivas: İmranlı-Zara 10. çeşmenin üzeri 1350 m km 1019-12 MYD 14.07.1997 6 P. integrifolia Hub.Mor. 7 8 9 10 11 12 13 14 15 16 17 18 Malatya: Malatya-Arapkir 30. km kuru çayı, derenin sağ yamaçları 770 m P. rigida LaBill. Malatya: Elbistan-Malatya 53 km Darende yol ayrımına 3 km kala 1600 m P. linearis Kayseri: Kayseri-K.Maraş Yedi Oluk mezarlık üzeri 1600 m P. rigida K.Maraş: Ahır dağı Yalnız Ardıç sırtları 1350 m P. linearis Kayseri: Aladağ maden yolu demir madeni Yahyalı 1700 m P. tuberosa Sivas: İmranlı-Zara 10. km çeşmenin üzeri 1350 m P. longifolia Boiss. & Hatay: Güzel Yayla (soğuk oluk) Blanche üzeri Çamlık tepe 1050 m P. samia L. Adana: Gülek Boğazı Gülek köyü – Adana 1. km 1100 m P. samia Adana: Gülek Boğazı Gülek köyü – Adana 1.km 1100 m P. longifolia Antalya: Hatay: Belen Kırcı köyü yakınları 650-750. m P. kotschyana Hub.- Antalya: Belen geçidi Top boğaz Mor. arası 550. m P. longifolia var. Hatay: St. Siman manastırı longifolia çevresi 550 m P. longifolia var. Hatay: Samandağ Yayladağı sahil longifolia yolu Çöğüntü-Gözene köyleri arası 270. m 21.06.1997 31.05.1997 1025 MYD 7.08.1997 1038 MYD 7.08.1997 1043 MYD 5.8.1997 1047-1 MYD 6.8.1997 1053 MYD 13.8.1997 1055-1MYD 16.8.1997 1058 MYD 14.9.1997 1060-1 MYD 13.9.1997 1060-3 MYD 13.9.1997 1061 MYD 15.9.1997 1066 MYD 13.06.1998 1069 MYD 14.06.1998 1070-3 MYD 14.06.1998 23 Tablo 2.1. (Devamı) 19 P. syriaca Boiss. 20 P. syriaca 21 P. armeniaca 22 P. pungens var. pungens 23 P. sieheana Rech. f. 24 P. samia 25 P. armeniaca 26 P. armeniaca 27 P. grandiflora var. grandiflora 28 P. carica 29 P. carica 30 P. grandiflora var. fimbrigera 31 P. pungens var pungens 32 P. rigida 33 P. sintenissii Rech. f. 34 P. oppositiflora 35 P. pungens var. hirta 36 P. sintenissii Gaziantep-Yeşilyurt altın üzüm arası 500 m Hatay, Hassa-Kırıkhan yolunun 33. km Fettahoğlu Taş ocağı sapağı karşısı 150-200 m Konya: Konya-Ereğli Karaman arası 70. km 1050 m Karaman: Ayrancı-Karaman arası Karaman’a 17 km kala 1050 m Karaman: Karaman-Ereğli çıkışı Karsa bisküvi çikolata fabrikası karşısı 1050 m Antalya: Antalya Korkuteli yolu Thermesos Milli Parkı yanı Hodrian çevresi 950 m Antalya: Korkuteli yolu Tahtalıbelini Korkuteli tarafına geçince sol yamaç 1000 m Antalya: Korkuteli-Antalya 20. Km 1000 m Antalya: Kale-Kaş arası Yavu Köyü çevresi 400-500 m. Denizli: Korkuteli-Denizli Yolu Fethiye yol ayrımını 2 km geçince Çavdır’a gelmeden 380 m Denizli: Korkuteli-Denizli Yolu Burdur-Teffeni yol ayrımını geçince Büyükalan-Küçükalan köyleri arası 1050 m Denizli: Afyon-Denizli Afyon yolu Çardak bozkırı 8.km Acıgöl’ün kuzeyi 850-950 m Burdur: Uluborlu Senirkent yolu Senirkent’e 8 km kala 950-1000 Malatya: Darende-Malatya 35. Km Elbistan yol ayrımına 3 km kala vericinin altı 1840 m Elazığ: Harput Buzluk mağarası Yolu Harput kalesi üzeri 1390 m Tunceli: Hozat-Pertek 13. Km askerin bulunduğu tepenin güne etekleri 1390 m Tunceli: Hozat-Pertek 11. Km Yenidoğdu Köyü sapağında 200 m içeride 1400 m Elazığ: Pekenik (Oymaağaç) Köyü Kırklar tepesi üzeri 1250 m 1079 MYD 15.06.1998 1078 MYD 15.06.1998 1088 MYD 24.06.1998 1089 MYD 24.06.1998 1090 MYD 24.06.1998 1105 MYD 26.06.1998 1107 MYD 26.06.1998 1109 MYD 26.06.1998 1115 MYD 27.06.1998 1123 MYD 28.06.1998 1124 MYD 28.06.1998 1127 MYD 29.06.1998 1129 MYD 29.06.1998 1138 F-2 MYD 16.07.1998 1141-B MYD 18.07.1998 1144 MYD 18.07.1998 1145 MYD 18.07.1998 1147-2 MYD 18.07.1998 24 Tablo 2.1. (Devamı) 37 P. capitata x kurdica 1150-A MYD 18.07.1998 1156 MYD 19.06.1995 1157 MYD 13.06.1998 1158 MYD 13.06.1998 1160 MYD 27.06.1998 1163-5 MYD 1163-7 MYD 1165 MYD 05.07.1998 44 P. fruticosa L. 1166 MYD 28.06.1998 45 1170 MYD 04.07.1998 1173 MYD 27.06.1998 38 P. armeniaca 39 P. amanica Vierh. 40 P. amanica 41 P. chimerae 42 P. samia (2 örnek) 43 P. pungens var. hirta 46 47 48 49 50 51 52 53 Malatya: Malatya-Venk köyü üzeri dağın yamaçları 1300 m Niğde: Çamardı Maden Mahallesi 1300 m Hatay: Arsus(Uluçınar) Haymaseki köyü sınırındaki eski krom maden ocakları Aktepe arkası 250-300 m Hatay: Arsus(Uluçınar) Haymaseki köyü sınırındaki eski krom maden ocakları Aktepe arkası 250-300 m Antalya: Kemer Kesmeboğazı 150 m Adana: Gülek boğazı Gülek Köyü-Adana 1. Km 1100 m Kayseri, Bünyan merkez Antalya: Kale Kaş arasındaki Elmalı yolundan Elmalı’ya giderken Gömüce Köyü-Kuruova arasındaki Oluk Yaylası 1700 m P. viscosa Poir. Adana: Saimbeyli-Feke 2. km Feke-Adana 930 m P. bourgaei Antalya: Antalya-Kemer Kesme boğazı 150 m P. amanica (2 örnek) Hatay: Arsus (Uluçınar) Haymaseki Köyü sınırındaki eski krom maden ocağı çevresi 250300 m P. viscosa Osmaniye: Düldül Dağı Kuşçu Köyünden Keklik Yaylasına çıkışta Düziçi (Haruniye) ilçesi 1100 m P. viscosa Osmaniye: Düldül dağı Kuşçu köyünden Keklik Yaylasına çıkışta Düziçi (Haruniye) ilçesi 1090 m P. viscosa Osmaniye: Yarpuz Yaylası Yolu 650 m P. viscosa Osmaniye: Yarpuz Yaylası Yolu 550 m P. kurdica x linearis Tunceli: Hozat Pertek 13. Km (2 örnek) askerin bulunduğu tepenin güney etekleri 1390 m P. linearis x Tunceli: Hozat Pertek 13. Km oppositiflora askerin bulunduğu tepenin güney etekleri 1390 m 06.07.1995 1179-3 MYD 19.05.1999 1179-8 MYD 1184 MYD 07.08.1998 1186 MYD 07.08.1998 1188 MYD 06.08.1998 1190 MYD 06.08.1998 1197 MYD 1199 18.07.1998 MYD 1201 MYD 18.07.1998 25 Tablo 2.1. (Devamı) 54 P. grandiflora var. fimbrilligera 55 P: lunariifolia x monocephala 56 P. leucophrocta 57 P. monocephala 58 P. pungens var. hispida K. Koch 59 P. bourgaei x chimerae Antalya: Anamur-Gazipaşa arası Kargıdan Çayı kıyısı mezarlık içi 20 m İçel: Aydıncık-Gülnar 13. Km 500 m İçel: Aydıncık-Gülnar 13. Km 500 m İçel: Gülnar Silifke 47. km 680 m 1225 MYD 07.06.1999 1226 MYD 07.06.1999 1228 MYD 07.06.1999 1231 MYD 07.06.1999 İçel: Gülnar Silifke 47. km 960 m 1236 MYD 07.06.1999 Antalya: Antalya-Kemer Ağva çayı kıyısı Batı kıyısındaki doğu yamaçlar 60 m 60 P. carica Denizli: Acıpayam-Denizli yolu Serinhisar’dan sonra 30. Km 1160 m 61 P. armeniaca x nisolii Burdur: Gülhisar Çavdar 15. Km Çavdar sapağından Gülhisar tarafına 1 km kala çeşmebaşı 1050 m 62 P. armeniaca Denizli: Sarayköy sapağı Babadağ 8. Km (Bekirler köyüne gelmeden) Boludüzü-Müze mıntıkası 450 m 63 P. armeniaca x carica Denizli: Sarayköy sapağı Babadağ 8. Km (Bekirler köyüne gelmeden) Boludüzü-Müze mıntıkası 450 m 64 P. carica Denizli: Sarayköy sapağı Babadağ 8. Km (Bekirler köyüne gelmeden) Boludüzü-Müze mıntıkası 450 m 65 P. bourgaei Muğla: Kale 31. Km 970 m 66 P. bourgaei x grandiflora (3 örnek) 67 P. lycia 68 P. grandiflora x lycia 69 P. pungens var. hispida 70 P. bruguieri Muğla: Kale 31. Km 970 m 1240 MYD 07.06.1999 1252 MYD 09.06.1999 1257 MYD 09.06.1999 1263 MYD 10.06.1999 1264 MYD 10.06.1999 1266 MYD 10.06.1999 1272 MYD 10.06.1999 1274 MYD 1275 10.06.1999 MYD 1276 MYD Muğla:Muğla-Kale 52. Km 1280 MYD 10.06.1999 virajda Kaleye giderken bayır çıkılıyor yüksek kayaların başı 950-1000 m Muğla: Muğla-Kale 52. Km 1282 MYD 10.06.1999 virajda Kaleye giderken bayır çıkılıyor yüksek kayaların başı 950-1000 m K.Maraş-Göksun geçince tünele 1284 MYD 16.06.1999 73. Km 1340 m Gaziantep: Soft Dağı tv vericisi 1287-3 MYD 16.06.1999 altı 1350-1450 m 26 Tablo 2.1. (Devamı) 1278 MYD 16.06.1999 1331 MYD 26.6.1999 1334 MYD 02.07.1999 1335 MYD 02.07.1999 1336 MYD 02.07.1999 1302 MYD 02.07.1999 1349 MYD 03.07.1999 1363 MYD 03.07.1999 1367 MYD 03.07.1999 1371 MYD 03.07.1999 1382 MYD 03.07.1999 1414 MYD 03.07.1999 1415 MYD 03.07.1999 1420 MYD 03.07.1999 1434 MYD 20.7.1999 1437 MYD 20.7.1999 87 P. lanceolata Muğla: Muğla-Kale 52. Km virajda kaleye giderken bayır çıkılıyor 950-1000 m Konya: Konya-Antalya il sınırı Beyşehir-Akseki yolunu 50. km Şakiroğlu çeşmesi 1200 m Sivas: Gürün-Sivas 24. Km Otlu kilise Demir Madeni İşletmesi sapağının karşısı 1850 m Sivas: Gürün-Sivas 24. Km Otlu kilise Demir Madeni İşletmesi sapağının karşısı 1850 m Sivas: Gürün-Sivas 24. Km Otlu kilise Demir Madeni İşletmesi sapağının karşısı 1850 m K.Maraş, Ilıca-Zorkun Yaylası arası ziyaret mevkiindeki soğuk suyun doğusu 1200 m Tokat: Turhal Hastane Tepesi (Devlet Hastanesi Doğu Yamaçları) 650 m Kayseri: Develi-Kayseri 8-9 Km 1720 m Kayseri: Develi-Kayseri 8-9 Km 1720 m Kayseri: Develi-Kayseri 8-9 Km 1720 m Kayseri: Develi-Kayseri birkaç km Kayseriye doğru 1550-1600 m Kayseri: Develi-Kayseri birkaç km Kayseriye doğru 1550-1600 m Kayseri: Develi-Kayseri birkaç km Kayseriye doğru 1550-1600 m Kayseri: Develi-Kayseri birkaç km Kayseriye doğru 1550-1600 m Van: Van Kalesi kuzey sınırlarının dış kenarları 1730 m Van: Çatak-Van 3. Km Kunispik suyu – Çatak arası 1670 m Van: Gürpınar 8 km. 2100 m 1438 MYD 20.7.1999 88 P. tuberosa Van: Çatak 20. Km 2210 m 1441-2 MYD 20.7.1999 89 P. armeniaca x nissollii Kayseri: Develi-Kayseri 5. Km 1446 MYD Dağ yolu 1680 m 71 P. grandiflora 72 P. grandiflora var. grandiflora 73 P. capitata 74 P. linearis 75 P. capitata x linearis 76 P. brunneogaleata 77 P. armeniaca 78 P. linearis 79 P. armeniaca x linearis 80 P. armeniaca 81 P. capitata x nissoli 82 P. kurdica x linearis 83 P. linearis x oppositiflora 84 P. capitata x kurdica 85 P. lanceolata Boiss. & Hohen. 86 P. lanceolata 08.08.1999 27 Tablo 2.1. (Devamı) 90 P. fruticosa İzmir: Çeşme Çiftlik Köyü 60-70 m 27.04.2000 107 P. lunariifolia x monocephala 108 P. leucophracta melezi 109 P. lunariifolia 1451-2 MYD 1451-3 MYD 1451-4 MYD K.Maraş: gişeler-Narlı 4. Km 1465 MYD Balkayası Çiçek Köyü civarı 600 m Van: Erciş Ulupamir Köyü köyün 1487 MYD batısı tepenin yamaçları Altındere arasının ilerisi 1730+ m İzmit: Maşukiye Köyü Kirazlı 1497-7 MYD yayla Muğla: Marmaris Karacaköy 1503 MYD Ovacık mah. 234 m Muğla: Marmaris Karacaköy 1504 MYD Ovacık mah. 234 m Muğla: Marmaris Karacaköy 1505 MYD Ovacık mah. 211 m Muğla: Marmaris Bozburun 1507 MYD Marmaris 10. Km tepe üstü 221 m Mugla: Marmaris Bozburun 1508 MYD Sercelimanı’nı çevreleyen tepeler 71 m Antalya: Kemer Ağvaderesi’nin 1522 MYD batı kıyıları 32 m Antalya: Kemer Ağvaderesi’nin 1524 MYD batı kıyıları 32 m Antalya: Alanya Kalesi’nin 1526 MYD doğusu dış ile iç sur arası 145 m Antalya: Alanya Kalesi’nin 1527 MYD doğusu dış ile iç sur arası 145 m Antalya: Alanya Kalesi’nin 1529 MYD doğusu dış ile iç sur arası 145 m 1530 MYD Antalya: Gazipaşa Kaledıran 1537 MYD Çayı (Kaledıran) 28 m İçel: Aydıncık-Gülnar 13. Km 1538 MYD 537 m İçel: Aydıncık-Gülnar 13. Km 1539 MYD 537 m İçel: Aydıncık-Gülnar 13. Km 1541 MYD 537 m İçel: Aydıncık-Gülnar 13. Km 1542 MYD 537 m Antalya: Gazipaşa Kaledran 28 m 1535- 3 MYD 110 P. kurdica K.Maraş: Narlı gişeler 650 m 15.06.2001 91 P. syriaca 92 P. lanceolata 93 P. russeliana 94 P. bourgaei 95 P. lycia x bourgaei 96 P. grandiflora x lycia 97 P. bourgaei 98 P. lycia 99 P. chimerae 100 P. bourgaei x chimerea 101 P. lunariifolia 102 P. leucophracta 103 P. leucophracta x lunariifolia 104 P. lunariifolia 105 P. monocephala 106 P. lunariifolia 1564 MYD 01.07.2000 19.07.1999 19.07.1999 05.05.2001 05.05.2001 05.05.2001 05.05.2001 06.05.2001 08.05.2001 05.05.2001 09.05.2001 09.05.2001 09.05.2001 09.05.2001 09.05.2001 09.05.2001 09.05.2001 09.05.2001 09.05.2001 28 Tablo 2.1. (Devamı) 111 P. brunneogaleata K.Maraş: Narlı gişeler 650 m 1568-2 MYD EY 15.06.2001 112 P. capitata x P. brunneogaleata var. brunneogaleata 113 P. capitata x brunneogaleata 114 P. bruguieri x brunneogaleata 115 P. bruguieri x capitata 116 P. capitata x kurdica Gaziantep: Soft Dağı 1456 m 1585 MYD 15.06.2001 Gaziantep: Soft Dağı 1456 m 1581 MYD 15.06.2001 Gaziantep: Soft Dağı 1456 m 1589 MYD 15.06.2001 Gaziantep: Soft Dağı 1456 m 1595 MYD 15.06.2001 Malatya: Venk Köyü 1230 m 1600 MYD 16.06.2001 117 P. kurdica Malatya: Venk Köyü 1230 m 1602 MYD 16.06.2001 118 P. pungens var. pungens 119 P. linearis Kayseri: Bünyan yolu Kaykop 1616-2 MYD doğusu eski askeri alan Sivas: Sızır Çat Ormanları 1617-2 MYD 14.06.1995 120 P. sieheana Rech. f. Kayseri: Gesi İldem Koop. karşısı 1150 m Mersin: Mut-Silifke yolu Gülnar sapağı kenarı 166 m İçel: Aydıncık Aydıncık – Gülnar 13. Km 520 m İçel: Aydıncık Aydıncık – Gülnar 13. Km 520 m İçel: Aydıncık Aydıncık – Gülnar 13. Km 520 m Antalya: Kemer-Ağva Çayı ~5 km 60m İçel: Aydıncık, Aydıncık– Gülnar 13. Km 520 m Konya: Doğanhisar sapağı – Konya 1. Km Şarkikaraağaç Konya arası 1148 m Antalya: Kemer-Ağva Çayı ~5 km 60m Kayseri: Yahyalı Maden yolu 1643 MYD 19.06.2003 1668 MYD 03.07.2003 1670 MYD 03.07.2003 1671-1 MYD 03.07.2003 1675 MYD 03.07.2003 1676 MYD 03.07.2003 1678 MYD 03.07.2003 1685 MYD 06.07.2003 1677 MYD 06.07.2003 1689 MYD 08.07.2003 Kayseri: Yahyalı Maden yolu 1690 MYD 1538 m Kayseri: Yahyalı Maden yolu 1694 MYD 1428 m Kayseri: Kayak evi Develi yolu 1698 MYD 13. Km den sağ taraf 2. km Adaca tepesi etekleri 1700 m 08.07.2003 121 P. nissolii 122 P. leucophracta 123 P. lunariifolia 124 P. leucophracta melezi 125 P. bourgaei x chimerae 126 P. bourgaei 127 P. pungens var. pungens 128 P. chimerea 129 P. capitata 130 P. pungens 131 P. capitata x nissolii 132 P. armeniaca 22.07.2001 08.07.2003 10.07.2003 29 Tablo 2.1. (Devamı) 133 P. kurdica 134 P. rigida 135 P. integrifolia 136 P. sintenisii 137 P. russeliana 138 P. amanica 139 P. longifolia var. bailanica 140 P. syriaca 141 P. viscosa 142 P. bruguieri 143 P. brunneogaleata var. brunneogaleata 144 P. brunneogaleata 145 P. nissolii 146 P. bruguieri x capitata 147 P. capitata x brunneogaleata var. brunneogaleata 148 P. capitata Malatya: Darende 35. Km K.Maraş Elbistan yol ayrımının 3 km vericinin altı batısı 1816 m Malatya: Darende 35. Km K.Maraş Elbistan yol ayrımı 3. km vericinin altı batısı 1816 m Malatya: Arapgir Sapağı 36. Km Kurucaydan 6 km ilerde 752 m Elazığ: Harput Mezarlık çevresi 1484 m Bolu: Abant Gölü çevresi 1220 m 1703 MYD 11.07.2003 1705 MYD 11.07.2003 (albino) 1706 MYD 1707 MYD 11.07.2003 1708 MYD 12.07.2003 1718-1 MYD 12.08.2003 Hatay: Arsuz, Haymaseki Köyü eski Maden ocağı çevresi 250300 m Hatay: Samandağ–Yeditepe 10. Km Karaköse Köyü 300 m Osmaniye, Ceyhan Yılankale yolun kenarı Osmaniye: Düziçi Çotlu köyü çevresi Berke barajı yolu Kuşçu Köyünden 3 km geride 520 m Gaziantep: Soft dağı Tv alıcısı çevresi 1450 m Gaziantep: Organize sanayi eski Adana yolu 25. Km Kızılyazı mevkii 955 m K.Maraş: Otoyol çıkışı Narlı 4. Km 600 m K.Maraş: Otoyol çıkışı Narlı 4. Km 600 m Gaziantep: Soft Dağı 1450 m 1723-1 MYD 22.05.2004 1724 MYD 22.05.2004 1727 MYD 23.05.2004 1728-1 MYD 23.05.2004 1732 MYD 07.06.2004 1733 MYD 07.06.2004 1735-1 MYD 07.06.2004 1739 MYD 07.06.2004 1744-1 MYD 24.06.2004 Gaziantep: Soft Dağı 1450 m 1749 MYD 24.06.2004 Gaziantep: Soft Dağı 1450 m 1750 MYD 24.06.2004 1751-2 MYD 24.06.2004 1817 MYD 07.07.2005 1818 MYD 07.07.2005 149 P. brunneogaleata var Gaziantep: Soft Dağı 1450 m brunneogaleata 150 P. capitata K.Maraş: Ahir Dağı tv verici altı ağaçlandırma sahası 1522 m 151 P. capitata x nissolii K.Maraş: Ahir Dağı tv verici altı ağaçlandırma sahası 1522 m 152 P. rigida Ermenek: Başyayla- Taşkent Karayolu 1880 m 1837 (albino) MYD 10.07.2005 30 Tablo 2.1. (Devamı) 153 P. pungens var hirta 154 P. pungens var. hispida 155 P. nissolii 156 P. russeliana 157 P. linearis 158 P. tuberosa 159 P. pungens x nissolii 160 P. nissolii 161 P. pungens var. hispida 162 P. nissolii 163 P. oppositiflora 164 P. physocalyx 165 P. capitata 166 P. oppositiflora x kurdica 167 P. physocalyx x oppositiflora 168 P. oppositiflora 169 P. kurdica x oppositiflora 170 P. kurdica 171 P. longifolia Çankırı: Kurşunlu Çerkez 4. Km 1195 m Kayseri: Yahyalı Maden yolu Dikme-Sazak Köyleri arası 1276 m Kayseri: Yahyalı Maden yolu Maden yıkama bölgesi 1553 m Amasya: Akdağ Taşlı Yayla Ladik yolu 5. Km Güneybati yamacı Karaçam altı 1633 m Bayburt: Aşkale yolu Kop geçidi Abide dibi 2370 m Van: Gedikbaşı Köyü çevresi 2390 m Kayseri: Yahyalı Maden yolu Zamantı üzerinde ilk köprü çevresi 1198 m Kayseri: Yahyalı Maden yolu Zamantı üzerinde ilk köprü çevresi 1198 m Kayseri: Yahyalı Maden yolu Zamantı üzerinde ilk köprü çevresi 1198 m Kayseri: Zamantı KöprüsüKayseri 11. Km çeşmenin karşısı 1517 m Sivas: Gürün-Pınarbaşı 24. Km Osmandede Köyü çeşme başı 1844 m Sivas: Gürün-Pınarbaşı 24. Km osmandede köyü çeşme başı 1844 m Malatya: Venk Köyü İlkOkulun altı 2. Km 1131 m Malatya: Venk Köyü İlkOkulun altı 2. Km 1131 m Sivas: Gürün-Pınarbaşı 24. Km Osmandede Köyü çeşme başı 1844 m Malatya: Venk Köyü, ilkokulun altı 1206 m Malatya: Venk Köyü, İlkOkulun altı 1206 m Malatya, Venk Köyü, İlkOkulun altı 1206 m Hatay: Belen Radar yolu tv vericisi yol ayrımı 1450 m 1853 MYD 15.07.2005 1858 MYD 23.07.2005 1861 MYD 23.07.2005 1863 MYD 1863-2 MYD 1863-3 MYD 1875-2 MYD 25.07.2005 1890-1 MYD 31.07.2005 1925-1 MYD 14.06.2008 1926-6 MYD 14.06.2008 1927-7 MYD 14.06.2008 1932-7 MYD 14.06.2008 1961 MYD 06.07.2008 1962 MYD 06.07.2008 1984-16 MYD 07.07.2008 1985.10-b MYD 1985.10-a MYD 1958-18 MYD 1958-24 MYD 07.07.2008 1988 MYD 07.07.2008 1985-25 MYD 07.07.2008 1989 MYD 07.07.2008 2008 08.07.2008 28.07.2005 06.07.2008 31 Tablo 2.1. (Devamı) 172 P. angustissima Hub.-Mor. Burdur: Salda Gölü çevresi 1200 m 173 P. monocephala 174 P. leucophracta 175 P. grandiflora var. grandiflora 176 P. armeniaca x nissolii 177 P. chimerae 178 P. lycia x bourgaei 179 P. pungens var. hirta 180 P. angustissima 2009 24.07.1996 Burdur: Gündoğmuş -Eğrigöl 10. Km 1350 m Isparta: Gündoğmuş egrigöl 10. Km 1350 m Antalya: Antalya’nın 10 km batısı 100500 m Kayseri: Erciyes Dağı, Develi üzeri Erciyes Hatıra Ormanı 1695 – 1800 m 6250 21.07.1996 6251 21.07.1996 2649-4 21.07.1996 2736 23.07.1996 Antalya: Antalya’nın 10 km batısı 100500 m Antalya: Antalya’nın 10 km batısı 100500 m Kayseri: Develi Bakırdağ– Saimbeyli 8. Km 1350 m Burdur: Dirmil Geçidi Serpantin yamaçları 1650 m 4904 25.07.2000 4905 25.07.2000 6327 30.07.2000 6261 22.07.2000 2.1.2. Kullanılan Kimyasal Maddeler ve Çözeltiler 2.1.2.1. DNA İzolasyonunda Kullanılan Kimyasal Maddeler ve Çözeltiler Bitki materyallerinden DNA izolasyonu aşamasında kullanılan kimyasal maddeler ve çözeltiler Tablo 2.2‘de verilmiştir. Tablo 2.2. DNA izolasyonunda kullanılan kimyasal maddeler ve çözeltiler KULLANILAN KİMYASAL MİKTAR -İÇERİK CTAB %2 100 mMTris 20 mM EDTA 1.4 M NaCl ve pH : 8 CTAB; %2’ lik 650 μl, Askorbik asit; %20’ lik, 30 μl Sodyum Bisülfit; %20’ lik, 60 μl ß-merkaptoetanol; %100’ lük, 6 μl ProteinazK 1mg/ml, 60 μl PVP; %20’ lik, 30 μl %20 2 g/10 ml CTAB tamponu Askorbik asit Sodyum Bisülfit ProteinazK çözeltisi ß-merkaptoetanol Kloroform – izoamil alkol Rnaz A çözeltisi İzopropanol % 70’ lik alkol % 95’ lik alkol Tris-EDTA Tamponu %20 2 g/ 10 ml 1 mg/ml Saf 24:1 10 mg/ml 2:3 oranında, DNA nın çöktürülmesinde DNA’nın yıkanmasında DNA’nın yıkanmasında 1x 10 mMTris 1 mM EDTA (pH: 8) 32 2.1.2.2. PCR Reaksiyonlarında Kullanılan Kimyasal Maddeler ve Çözeltiler Örneklerin ISSR bölgesine özgü polimeraz zincir reaksiyonunda kulanılan kimyasal maddeler ve çözeltiler Tablo 2.3‘te verilmiştir. Tablo 2.3. Polimeraz Zincir Reaksiyonlarında kullanılan kimyasal maddeler ve çözeltiler PCR su (Sigma) 12,75 µl 10x Tampon (Fermentas) 2,5 µl 1000 dNTP (Fermentas) 3,75 µl 100 µm Primer (Operon) 0,5 µl 25 mM MgCl2 (Fermentas) 2 µl Genomik DNA 2 µl TaqPolimeraz – 5 u/ml 0,2 µl (1 ü. ISSR) Reaksiyon Hacmi 23,7 µl (ISSR) 2.1.2.3. Agaroz Jel Elektroforezinde Kullanılan Kimyasal Maddeler ve Çözeltiler PCR ürünlerinin görüntülendiği agaroz jel elektroforezinde kullanılan kimyasal maddeler ve çözeltiler Tablo 2.4’te verilmiştir. Tablo 2.4. Agaroz jel elektroforezinde kullanılan kimyasal maddeler ve çözeltiler Kullanılan Kimyasallar Agaroz Jel TBE (Tris, Borat, EDTA) DNA ladder Etidyum Bromür çözeltisi Yükleme Boyası Miktar ve İçerik %1,5 1,5 g Agaroz 100 ml TBE 1X pH: 8 21.8 g Tris 1.17 g EDTA 21-21.5 g Borik Asit 500 ng/µl 5 mg/ml 6X 33 2.1.3. Primerler Bu çalışmada kullanılan primerlerin baz dizileri, % G+C içerikleri, Tm sıcaklıkları Tablo 2.5’te verilmiştir. Tablo 2.5. ISSR bölgesini amplifiye etmede kullanılan primerler 810 UBC Primer Baz Dizisi (5’-3’) (GA)8T Primerin %G+C içerikleri 47,06 PrimerinTm Sıcaklığı (0C) 48 811 (GA)8C 52,94 50 814 (CT)8A 47,06 48 815 (CT)8G 52,94 50 818 (CA)8G 52,94 50 822 823 (TC)8A (TC)8C 47,05 52,94 48 50 824 (TC)8G 52,94 50 826 (AC)8C 52,94 50 827 (AC)8G 52,94 50 835 (AG)8YC 50 – 55,56 52 836 840 (AG)8YC (GA)8YT 50 – 55,56 44,4 - 50 52 52 841 (GA)8YT 44,4 - 50 52 842 (GA)8YG 50 – 55,56 54 844 (CT)8RA (CA)8RG 44,4 - 50 50 50-55,56 54 Primer Adı 848 2.2. Metot 2.2.1. Morfolojik İncelemeler Farklı lokalitelerden toplanmış 36 adet Phlomis taksonu ve 46 adet Phlomis melezi vertisillat sayısı, vertisillatlar arası uzunluk taban, gövde ve çiçek yapraklarının eni, boyu ve petiyol uzunlukları gibi toplamda 24 karakter esas alınarak morfolojik ölçümleri yapılmıştır. Herhangi bir nedenle bir veya daha fazla yapısı eksik olan 34 örneklerin o özelliği değerlendirmeye alınmamıştır. Çalışmadaki örneklerin ve morfolojik karakterlerin sayıca çokluğu göz önüne alınarak bu verilerin daha iyi gözlenebilmesi için morfolojik karakterler harflerle kodlanmıştır (Tablo. 2.6). Örneklerin morfolojik ölçüm sonuçları Ek 1’de verilmiştir. Tablo 2.6. Morfolojik karakterler ve kodlamaları Kod Karakter A Vertisillat sayısı B Vertisillatlar arası uzunluk(cm) C Çiçek sayısı(1.Vertisillat ) D Çiçek sayısı(2.Vertisillat) G Çiçek sayısı(3.Vertisillat) H Taban Yaprağı boyu max.(cm) I Taban Yaprağı eni max.(cm) J Taban Yaprağı petiyol uzunluğu max.(cm) K Gövde Yaprağı boyu max.(cm) L Gövde Yaprağı eni max.(cm) M Gövde Yaprağı petiyol uzunluğu max.(cm) N Çiçek Yaprağı boyu max.(cm) O Çiçek Yaprağı eni max.(cm) P Çiçek Yaprağı petiyol uzunluğu max.(cm) Q Kaliks uzunluğu (cm) R Kaliks eni (cm) S Kaliks dişlerinin uzunluğu min.(cm) T Brakteol uzunluğu (cm) U Korolla uzunluğu(cm) V W X Y Z Korrolla üst dudak düz (1) eğik (2) Koralla alt dudak birbirine yakın büyüklükte (1) ortadaki büyük yanlardaki küçük (2) Bitki otsu (1) Brakteol sert bükülmez (1) çalı (2) yumuşak bükülebilir veya yok (2) Korolla rengi pembe mor (1) sarı (2) Alt dudak sarı üst dudak kahverengi(3) Alt dudak pembe mor üst dudak sarı (4) beyaz (5) 35 Morfolojik ölçüm verileri NTYS v 2.11 [70] ve Splittree4 v 4.13.1 [71] programlarına aktarılarak değerlendirme yapılmıştır. Morfolojik verilere dayalı phylogram ağacı ve network grafikleri oluşturulurken Manhattan mesafe değerleri (Manhat) yöntemi kullanılmıştır. 2.2.2. Moleküler İncelemeler Erciyes Üniversitesi Biyoloji Bölümü herbaryumunda korunan örneklerin yapraklarından DNA izolasyonu yapılmış, örneklerin ISSR bölgeleri 17 farklı primer ile amplifiye edilmiştir. Amplifikasyon ürünleri agaroz jel elektroforezi ile yürütülmüş, ethidyum bromür ile boyanarak UV altında görüntülenmiştir. Elde edilen bant profilleri varlık/yokluk ve polimorfizm açısından değerlendirilmiştir. Moleküler veriler NTYS ve Splittree4 programlarına aktarılarak phylogram ağacı ve network grafiği oluşturulmuştur. 2.2.2.1. DNA İzolasyonu Herbaryumdan temin edilien, her bir türe ve meleze ait bitki yaprakları porselen havanda sıvı azot ile ezilerek toz haline getirilmiştir. Ezilen bitki örneklerinin yaprakları etiketlenmiş tüpler içerisinde -20˚C’de muhafaza edilmiştir. DNA izolasyonu toz hale getirilmiş bu materyallerden Yüzbaşıoğlu ve Dadandı’da belirtildiği şekilde yapılmıştır [35]. Genomik DNA izolasyonu protokolü: 1.5 µl'lik eppendorf tüplerde Tablo 2.2’de belirtilen CTAB tampon hazırlanmış, üzerine 40 µg ezilmiş bitki örneği eklenmiştir. Karışımın homojen olarak dağılması için tüpler sallanmış ve önceden 60˚C'de hazırlanmış su banyosuna konulmuştur. Tüpler 15 dakikada bir ters düz edilerek 45 dk su banyosunda tutulmuştur. Üzerlerine 24/1 Kloroform izoamil alkol karışımın 2/3'ü oranında eklenmiş ve tüpler 50 defa ters düz edilmiştir. Kırılmış buz üzerinde 10–30 dk bekletilen tüpler ardından 14000 devirde +4˚C'de 6 dk santrifüj edilmiştir. Santrifüjden alınan tüplerdeki süpernatant kısmı alınmış, üzerlerine tekrar 2/3 oranında 24:1 kloroform-izoamil alkol eklendikten sonra tekrardan buz üzerinde 10–30 dk bekletilmiştir. Tüpler tekrar santrifüj edilmiş, süpernatant kısmı alınmıştır. Üzerlerine 5-6 µl RNAaz eklenmiş ve tüpler enzimin tüp 36 içerisinde dağılması için bir kaç kez nazikçe sallandıktan sonra 37.5˚C su banyosunda 1 saat bekletilmiştir. Sıcak su banyosundan alınan tüplerin üzerine 2/3 oranında izoprapanol eklenmiş, tüpler 4–5 defa sallanıp +4˚C’de gece boyu bekletilmiştir. Bekleme süresinin ardından tüpler 14000 devirde 2 dk +4˚C’de tutulmuştur. Tüplerin diplerinde kümelenmiş DNA yığınlarına dikkat edilerek süpernatant dökülmüş, tüplerin üzerine %70’lik alkol konulup tüpler ters çevirilerek iyice temizlenerek yıkanmıştır. Tüplerin üzerine yine aynı şekilde %70'lik alkol eklenmiş ve yarım saat bekletilmiştir. Yarım saat sonunda bu işlem %95'lik alkolle tekrarlanmıştır. Son yıkaması tamamlanan tüpler oda sıcaklığında bir gece kurumaya bırakılmıştır. Kuruyan DNA'ların üzerine 160µl (tüplerin dibinde birikmiş DNA miktarına göre bu miktar artırılmış ya da azaltılmıştır) 1X TrisEDTA solüsyonu eklenmiş ve çözünmeye bırakılmıştır. Çözünme işlemi tamamlandığında örnekler bir sonraki işlem için +4˚C’de muhafaza edilmiştir. 2.2.2.2. DNA konsantrasyonlarının belirlenmesi ve muhafaza edilmesi DNA’nın konsantrasyonu ve kalitesi bir sonraki işlemlerde sağlıklı sonuçlar elde edebilmek için oldukça önemlidir. Genomik DNA izolasyonu aşamasının başarısını test etmek ve elde edilmiş olan DNA’ların konsantrasyonlarını kontrol etmek amacıyla %1’lik agaroz jel hazırlanmıştır. Örneklerden elde edilen DNA’lar 1X TBE tamponu kullanılarak yürütülmüştür. Herhangi bir sorun tespit edilen örneklerde izolasyon aşaması tekrarlanmıştır. 2.2.2.3. Primer seçimi ISSR bölgesine yönelik UBC serisi 17-18 bazlık 28 primer denenmiştir. Bu primerlerden 11 tanesi amplifikasyon başarısı bakımından, 5 tanesi bant profili kalitesi bakımından yeterli bulunmamıştır. Çalışma 17 ISSR primeriyle tamamlanmıştır. 2.2.2.4. ISSR reaksiyonları ISSR reaksiyonlarının başarılı bir şekilde gerçekleştirilebilmesi için PCR karışım konsantrasyonları, reaksiyonların gerçekleşecek olduğu sıcaklık ve süre değerleri optimize edilmiştir. ISSR reaksiyonlarında 2 ml’lik ince cidarlı tüpler kullanılmış ve 37 son hacim 23,7 µl olacak şekilde hazırlanmıştır. ISSR reaksiyonlarının gerçekleştiği sıcaklık ve süre değerleri Tablo 2.7’de verilmiştir. Tablo 2.7. ISSR-PCR döngüsü ADI SICAKLIĞI SÜRESİ Ön - Denatürasyon 940C 2dk/1 döngü Denatürasyon 940C 1dk/45 döngü 48-540C 1dk/45 döngü Uzama 720C 2dk/45 döngü Son – uzama 720C 5dk/1 döngü Bekleme 40 C - Bağlanma 2.2.2.5. Agaroz jel elektroforezi ve bantların boyanması ISSR bölgesinin amplifikasyonu tamamlanmış örneklerinin PCR ürünlerini ayrıştırmak için %1,5 agaroz jeli hazırlanmıştır. Brom-fenol-blue katılan amplifikasyon ürünleri hazırlanan agaroz jele yüklenmiş ve 85 V / 135 dk. yürütülmüştür. Ayrıştırılan ISSR bantları etidyum bromür ile boyanarak görüntüleme sisteminde fotoğrafları çekilmiştir. 2.2.2.6. ISSR bantlarının büyüklüğü ISSR bantlarının uzunluğunu tespit etmek için her bir agaroz jeline DNA ladder yüklenmiştir. Bu çalışmada Vivantis firmasına ait 100- 3000 bç arasında 15 fragmente ayrışan DNA ladder kullanılmıştır. 2.2.2.7. ISSR verilerinin değerlendirilmesi PCR da çoğaltılan ISSR ürünleri agoroz jellerde ayrıştırılmış ve jellerdeki bant profilleri İmage Analiz sisteminde fotoğraflanmıştır. Fotoğraflar bantların varlığı ve büyüklüğü açısından incelenmiştir. ISSR bantlarının varlığı “1” yokluğu ise “0” olarak kaydedilmiştir. Elde edilen skorlama verileri NTYS programında değerlendirilerek benzerlik indeksleri bulunmuştur. Bu aşamadan sonra SAHN programı kullanılarak kümelenme oluşturularak filogram ağacı (UPGMA) görüntülenmiştir. Filogram ağacı 38 Dice benzerlik indeksine göre oluşturulmuştur. Buna ek olarak veriler Splittree4 programına yüklenerek network grafiği oluşturulmuştur. Network grafiği ise 1’den Dice benzerlik indeksinin çıkarılmasıyla elde edilen genetik mesafe değerleri üzerine kurulmuştur. 39 3. BÖLÜM BULGULAR 3.1. Morfolojik Bulgular Türkiye’nin farklı bölgelerinden toplanan 46 adet Phlomis melezi ve 36 Phlomis taksonuna ait 193 örnek, 24 kantitatif karakter göz önüne alınarak incelenmiştir. Bu morfolojik ölçümler Ek 1’de verilmiştir. Bu ölçümler sonucu elde edilen morfolojik veriler NTSYS versiyon 2.11 programında kullanılarak Manhattan mesafeleri bulunmuştur. Kümeleme aynı programın bir alt uygulaması olan SAHN (upgma) yöntemi kullanılarak yapılmıştır. Morfolojik verilerin phylogram ağacı Manhattan mesafeleri üzerine kurulmuştur (Şekil 3.1). Splitstree 4 versiyon 4.13.1 programında morfolojik veriler kullanılarak neighbour network oluşturulmuştur (Şekil 3.2). Network oluşturulurken Manhattan genetik mesafe formülünden elde edilen değerler kullanılmıştır. Morfolojik ölçüm verilerine bağlı olarak hesaplanan Manhattan mesafeleri üzerine kurulan phylogram ağacı yaklaşık 3.80 Manhattan mesafesinden kesilirse Phlomis taksonları iki ana grupta toplanabilir. Birinci ana grupta çoğunlukla Phlomis seksiyonunun Dendrophlomis alt seksiyonuna giren taksonların (18 örnek) örnekleri yer alırken Oxyphlomis alt seksiyonuna ait taksonların 3 örneği, Gymnophlomis alt seksiyonunun bir taksonu ve Phlomoides seksiyonuna ait Phlomis tuberosa türünün 5 örneği de bu grupta yer almıştır. İkinci ana grup 2.5 Manhattan mesafesinden kesilirse iki alt gruba ayrılabilir. Birinci alt grupta çoğunlukla Dendrophlomis alt seksiyonuna giren taksonların örnekleri (37 örnek) yer alırken, Oxyphlomis alt seksiyonuna ait taksonların 8 örneği ve Gymnophlomis alt seksiyonuna ait taksonların 7 örneği de bu grupta yer almıştır. İkinci alt grupta ise çoğunlukla Gymnophlomis alt seksiyonuna giren taksonların örnekleri (49 örnek) yer alırken, Oxyphlomis alt seksiyonuna ait taksonların 40 13 örneği ve Dendrophlomis alt seksiyonuna ait taksonların 3 örneği de bu alt grupta yer almıştır. Morfolojik ölçüm verilerine bağlı olarak hesaplanan Manhattan mesafeleri üzerine kurulan phylogram ağacı incelendiğinde Bentham’ın yaptığı [6] Phlomis seksiyonu Oxyphlomis (pembe–mor çiçekli ve otsu bitkiler; P. pungens P. samia P. integrifolia P. rigida), Gymnophlomis ( sarı çiçekli ve otsu; P. oppositiflora, P. armeniaca, P. nissolii, P. linearis, P. physocalyx, P. capitata, P. kurdica, P. bruguieri, P. angusstisima, P. kotshyana, P. sieheana, P. sintenisii, P. lanceolata, P. syriaca, P. carica, P. brunneogaleata) ve Dendrophlomis (odunsu; P. lycia, P. grandiflora, P. bourgaei, P. chimerae, P. lunariifolia, P. grandiflora, P. monocephala, P. leucophracta P. furuticosa P. russeliana P. viscosa P. amanica) olmak üzere 3 alt seksiyona ayıran gruplamaya uyum göstermediği görülmektedir. Çalışmada bulunan 36 farklı Phlomis taksonu bu gruplamaya uygun bir ayrım göstermemiştir (Şekil 3.1 ). Pholomoides seksiyonunun tek üyesi olan P. tuberosa (5 örnek ) taksonunun örnekleri filogenetik ağaçta birinci ana grupta Oxyphlomis ve Dendrophlomis alt seksiyonunun üyeleri ile birlikte gruplanmıştır. Çalışmada morfolojik karakterlerden elde edilen Manhattan mesafeleri kullanılarak oluşturulan filogenetik ağaç incelendiğinde Phlomis melezlerinin ebeveynlerine göre yerleşimi aşağıdaki kategorilere toplanabilir: (1) Melez ebeveynleri arasında yerleşmiştir. Bu yerleşme ebeveynlerden biriyle fakat kardeş takson olmayacak şekildedir (sister) (MYD 1275-1 kodlu P. bourgaei x grandiflora; MYD 1276 kodlu P. bourgaei x grandiflora; MYD 1415 kodlu P.linearis x oppositiflora; MYD 1818 kodlu P.capitata x nissolii). (2) Melez atalarından birine tabandan bağlanmıştır (1529 kodlu P. leucophracta x lunariifolia). (3) Melez atalardan birine kladistik olarak yakın bir şekilde yerleşmiştir (MYD 1201 kodlu P. linearis x oppositiflora; MYD 1504 kodlu P. bourgaei x lycia; MYD 1530 kodlu P. leucophracta x lunariifolia; MYD 2736 kodlu P. armeniaca x nissolii, MYD 1589 kodlu P. bruguieri x brunneogaleata; MYD 1985-25 kodlu P. kurdica x oppositiflora). (4) Melez diğer melezler/taksonlarla bir arada gruplanmıştır (MYD 1199 kodlu P.kurdica x linearis; MYD 1367 kodlu P. armeniaca x linearis; MYD 1420 kodlu P. kurdica x capitata; MYD 1150-A-1 kodlu P. kurdica x capitata; MYD 1226 kodlu P. lunarifolia x monocephala; MYD 1264 kodlu P. armeniaca x carica; MYD 1446 kodlu P. armeniaca x nissolii; MYD 1524 kodlu P. bourgaei x chimerae; MYD 1541 kodlu P. lunariifolia x monocephala; MYD 1568-2 41 kodlu P. kurdica x nissolii; MYD 1585 kodlu P. capitata x brunneogaleata; MYD 1581 kodlu P. capitata x brunneogaleata; MYD 1676 kodlu P.bourgaei x chimarea; MYD 1694 kodlu P. capitata x nissolii; MYD 1925-1 kodlu P. nissolii x pungens; MYD 1197 kodlu P. kurdica x linearis; MYD 1240 kodlu P. bourgaei x chimerae; MYD 1257 kodlu P. armeniaca x nissolii; MYD 1274 kodlu P. bourgaei x grandiflora; MYD 1282 kodlu P. grandiflora x lycia; MYD 1336 kodlu P.capitata x linearis; MYD 1382 kodlu P.capitata x nissolii; MYD 1414 kodlu P.kurdica x linearis; MYD 1505 kodlu P.grandiflora x lycia; MYD 1595 kodlu P.brugueri x capitata; MYD 1600 kodlu P.kurdica x capitata; MYD 1744-1 kodlu P.brugueri x capitata; MYD 1749 kodlu P.capitata x brunneogaleata; MYD 1985-10-B kodlu P.kurdica x oppositiflora; MYD 1958-18 kodlu P.physocalyx x oppositiflora; MYD 1985-10-A kodlu P.kurdica x oppositiflora; MYD 1958-24 kodlu P.physocalyx x oppositiflora; HD 4905 kodlu P.bourgaei x lycia; MYD 1542 kodlu P.leucophracta melezi; MYD 1589 kodlu P.brugueri x brunneogaleata; MYD 1585 kodlu P.capitata x brunneogaleata; MYD 1675 kodlu P. leucophracta melezi) (Şekil 3.1). Morfolojik veriler kullanılarak elde edilen neighbour network modelinin verilere uyumu oldukça yüksek olup, %98.56’dır (fit=90.2, LSfit= 98.56). Network grafiğinin topolojisi incelendiğinde network’ün merkezinde ve oluşan grupların içerisinde bir nedeni retikülat olaylar’da olan verideki uyumsuzlukları gösteren çok sayıdaki paralel kenarlardan oluşan kutucuklar yer almaktadır. Bu bulgular araştırmada kullanılan Phlomis örneklerinin uzak ve yakın tarihsel geçmişinde melezleşme gibi retikülat olayların yer aldığını göstermektedir. Morfolojik verilerden elde edilen network grafiğinin topolojisi incelendiğinde Phlomis türlerinin 2 ana grupta toplandığı görülmektedir. Birinci ana grupta çoğunlukla Gymnophlomis alt seksiyonunun örnekleri yer alırken, Dendrophlomis alt seksiyonunun üç örneği (MYD 1503 kodlu P. bourgaei, HD 6251 kodlu P. leucophracta, HD 6250 kodlu P.monocephala) ve Oxyphlomis alt seksiyonun yedi örneği (MYD 1060-4 ve 1105 kodlu P. samia, HD 6327, MYD 1690, MYD 1853 ve 1858 kodlu P.pungens, MYD 1707 kodlu P. integrifolia) de bu grupta yer almaktadır. Birinci grupta Gymnophlomis alt seksiyonunun P. armeniaca türünün MYD 1088, 1107, 1109, 1156, 1263, 1349, 1698 kodlu, P.carica türünün MYD 1123, 1124, 1252, 1266 kodlu örnekleri, P.linearis türüne ait MYD 1053, 1335, 1363, 1617-2, 1875-2 kodlu örnekleri, P. capitata türüne ait MYD 1750, 1817, 1984-16 kodlu 42 örnekleri, P. angusstisima türüne ait MYD 2009 kodlu, P.oppositiflora türüne ait MYD 1961, 1988, 1144 kodlu, P.physocalyx türüne ait MYD 1962 kodlu, P.kotshyana türüne ait MYD 1066 kodlu, P. sieheana türüne ait MYD 1090, 1643 kodlu, P. sintenisii türüne ait MYD 1141-B, 1708 kodlu, P. lanceolata türüne ait MYD 1434, 1437, 1438 kodlu, P. brunneogaleata türüne ait MYD 1302, 1733, 1735-1, 1751-2 kodlu, P. nissolii türüne ait MYD 1739, 1865, 1926-6, 1932-7 kodlu, P. syriaca türüne ait MYD 1078-1, 1079, 1465 kodlu, P. kurdica türüne ait MYD 1602, 1989 kodlu örnekleri yer almaktadır (Şekil 3.4). İkinci ana grupta çoğunlukla Dendrophlomis alt seksiyonunun örnekleri yer alırken (55 örnek), Oxyphlomis alt seksiyonunun 17 örneği (MYD 1706, 1038, 1047-1, 1837, 1138-F-2, 1705-1 kodlu P. rigida, MYD 1163-5, 1163-7 ve 1068 kodlu P.samia, MYD 1025 kodlu P.integrifolia, MYD 1616-2, 1089 kodlu P. pungens var pungens, MYD 1236, 19277 kodlu P. pungens var hispida, MYD 1284, 1165 ve 1145 kodlu P. pungens var hirta), Gymnophlomis alt seksiyonunun 12 örneği (MYD 1371 kodlu P.armeniaca, MYD 1732 kodlu P. bruguieri, MYD 1727 kodlu P. syriaca, MYD 1564 ve 1703 kodlu P. kurdica, MYD 1278 kodlu P. bruguieri, HD 6261 kodlu P. angustissima, MYD 1334 ve 1689 kodlu P. capitata, MYD 1043 kodlu P. linearis, MYD 1147-2 kodlu P. sintenisi, MYD 1668 kodlu P. nissolii) ve Phlomoides seksiyonunun tek türü olan P. tuberosa’nın 5 örneği (MYD 1441-2, 1019-4, 1890-1, 1055-1 ve 10199 kodlu) de bu grupta yer almaktadır. İkinci grupta Dendrophlomis alt seksiyonunun P. russeliana türüne ait; MYD 1497-7, 1718-1, 1863(3 örnek) kodlu, P. furuticosa türüne ait; MYD 1166, 1451(3 örnek) kodlu, P. lunariifolia türüne ait; MYD 1526, 1537, 1539, 1535-3, 1671-1kodlu, P. grandiflora türüne ait MYD 1115, 1127-B, 1225, 1278-1, 1331, 2649-4 kodlu, P. viscosa türüne ait MYD 1170, 1184-1, 1186, 1188-2, 1190-2, 1728-1 kodlu, P. bourgaei türüne ait MYD 1002, 1173, 1272, 1507, 1678 kodlu, P. leucophracta türüne ait MYD 1017-2, 1228-2, 1670 kodlu, P. longifolia türüne ait MYD 1058, 1061, 1069, 1070-3, 1724, 2008 kodlu, P. amanica türüne ait 1157-2, 1158-1, 1179(2 örnek), MYD 1723-1 kodlu, P. lycia türüne ait MYD 1006, 1280, 1508 kodlu, P. monocephala türüne ait MYD 1231, 1538 kodlu, P. chimerae türüne ait MYD 1160, 1522, 1677, 4904 kodlu örnekler yer almaktadır. Ayrıca, ikinci ana grupta Phlomoides seksiyonuna ait tek tür olan P. tuberosa’nın MYD 1441-2, 10194 ve 9, 1890-1, 1055-1 kodlu örnekleri de yer almaktadır (Şekil 3.5). 43 Morfolojik veriler kullanılarak Manhattan mesafesi formülünden elde edilen mesafe matriksi üzerine kurulan filogenetik network incelendiğinde melezlerin atalarına göre konumları şu gruplarda toplanabilir: (1) Melez atalarından birinin hemen yanında yer almaktadır (MYD 1985-25 kodlu P. kurdica x oppositiflora, MYD 1985-10-B kodlu P. kurdica x oppositiflora, MYD 1199 P. kurdica x linearis, MYD 1818 kodlu P. capitata x nissolii, 1367 kodlu P. armeniaca x linearis, MYD 1529 kodlu P. leucophracta x lunariifolia, MYD 1275-4 kodlu P. bourgaei x grandiflora, MYD 1541 kodlu P. lunariifolia x monocephala, MYD 1276 kodlu P. bourgaei x grandiflora). (2) Melez atalarının birisinin veya her ikisinin bulunduğu grupta yer almaktadır (MYD 1414 kodlu P. kurdica x oppositiflora, MYD 1985-10-A kodlu P. kurdica x oppositiflora, MYD 1150-A-1 kodlu P. kurdica x capitata, MYD 1420 kodlu P. kurdica x capitata, MYD 1568-2 kodlu P. kurdica x nissolii, MYD 1415 kodlu P. linearis x oppositiflora, MYD 1925-4 kodlu P. nissolii x pungens, MYD 1201 kodlu P. linearis x oppositiflora, MYD 1606 kodlu P. kurdica x capitata, MYD 1958-24 kodlu P. physocalyx x oppositiflora, MYD 1958-8 kodlu P. physocalyx x oppositiflora, MYD 1257 kodlu P. armeniaca x nissolii, MYD 1264 kodlu P. armeniaca x carica, MYD 1749 kodlu P. capitata x brunneogalata, MYD 1382 kodlu P. capitata x nissolii, MYD 1446 kodlu P. armeniaca x nissolii, MYD 1589 kodlu P. bruguieri x brunneogalata, MYD 1585 kodlu P. capitata x brunneogalata, MYD 1336 kodlu P. capitata x linearis, MYD 1675 kodlu P. bourgaei x chimerae, MYD 1676 kodlu P. bourgaei x chimerae, MYD 1524 kodlu P. bourgaei x chimerae, MYD 1530 kodlu P. leucophracta x lunariifolia, MYD 1505 kodlu P. grandiflora x lycia, MYD 1274 kodlu P. bourgaei x grandiflora, MYD 1504 kodlu P. bourgaei x lycia, MYD 1226 kodlu P. lunariifolia x monocephala, MYD 1282 kodlu P. grandiflora x lycia) (3) Melez atalardan birinin veya ikisininde yer almadığı grupta bulunmaktadır (MYD 2736 kodlu P. armeniaca x nissolii, MYD 1595 kodlu P. bruguieri x capitata, 1581 kodlu P. capitata x brunneogalata, MYD 1744 kodlu P. bruguieri x capitata, MYD 1694 kodlu P. capitata x nissolii, MYD 1197 kodlu P. kurdica x linearis, MYD 1542 kodlu P. leucophracta melezi, HD 4905 kodlu P. bourgaei x lycia, MYD 1240 kodlu P. bourgaei x chimerae). 44 Şekil 3.1. Phlomis örneklerinde morfolojik verilere ait phylogram ağacı (parça 1) 45 Şekil 3.2. Phlomis örneklerinde morfolojik verilere ait phylogram ağacı (parça 2) 46 Şekil 3.3 Phlomis örneklerinde morfolojik verilere ait phylogram ağacı (parça 3) 47 Şekil 3.4. Phlomis örneklerinin morfolojik verilerinin Manhattan mesafe değerlerine göre oluşturulmuş network analizi 1. Parça 48 Şekil 3.5 Phlomis örneklerinin morfolojik verilerinin Manhattan mesafe değerlerine göre oluşturulmuş network analizi 2. Parça 3.2 Moleküler Bulgular Morfolojik ölçümlerden sonra 193 örneğin DNA’ ları izole edilmiş ve seçilen 17 farklı ISSR primeri ile ampilifikasyonları yapılmıştır. Bazı Phlomis tür/melezlerine ait bantların agaroz jel görüntüsü Şekil 3.6 da verilmiştir. PCR da çoğaltıldıktan sonra 49 agaroz jelde yürütülen PCR ürünlerinin jel görüntüleri bantın varlık- yokluk durumuna göre sırasıyla 1 ya da 0 olarak skorlanmıştır. Şekil 3.6. 840 ISSR primeri ile ampilifikasyonu yapılan bazı DNA örneklerinin jel görüntüleri, Marker görüntüsü 17 farklı ISSR primerinden toplam olarak 415 bant profili elde edilmiştir. Elde edilen 415 bantın tamamı polimorfik bantlardır. Çalışma sonucunda hiç monomorfik bant elde edilmemiştir. Elde edilen 415 polimorfik bantın büyüklükleri 100-3000 baz çifti arasında değişmektedir. ISSR primerlerinin polimorfik bant büyüklükleri ve sayıları Tablo 3.1 de verilmiştir. Tablo 3.1 ISSR primerleri ile elde edilen polimorfik bant sayıları ve büyüklükleri Polimorfik bant sayısı 17 24 26 22 Bant büyüklüğü (baz çifti) 810 814 811 815 Toplam bant sayısı 17 24 26 22 818 822 823 824 24 21 23 26 24 21 23 26 100-900 200-1000 200-900 200-1200 826 827 835 836 840 841 27 28 24 27 24 28 27 28 24 27 24 28 150-1000 125-1500 100-1000 125-1200 125-2000 125-3000 842 844 848 23 23 28 23 23 28 125-900 125-1200 150-1300 ISSR primer 100-1500 200-1200 200-1500 200-1200 50 3.2.1 ISSR verilerine ait Phylogram ağacı Çalışmada kullanılan 193 örneğin ISSR verilerinin phylogram ağacı Dice benzerlik indeksine göre oluşturulmuştur. Phylogenetik ağaç oluşturulurken NTSYS versiyon 2.11 paket programı kullanılmıştır. NTSYS paket programı ile benzerlik indeksleri bulunduktan sonra kümelemede aynı programın bir alt uygulaması olan SAHN (UPGMA) programı kullanılmıştır. Phylogram ağacı Şekil 3.7, Şekil 3.8 ve Şekil 3.9’ da 3 parça halinde verilmiştir. ISSR verileri kullanılarak dice benzerlik indeksinden elde edilen benzerlik matriksi üzerine kurulan phylogram ağacı incelendiğinde 193 Phlomis örneği 2 ana grup altında toplanmıştır. Her bir ana grup kendi içerisinde iki alt gruba ayrılmıştır. Phylogram ağacı incelendiğinde Phlomis seksiyonunun altseksiyonu olan Dendrophlomis, Gymnophlomis ve Oxyphlomis taksonlarının örnekleri ve Phlomoides seksiyonunun tek türü olan Phlomis tuberosa’ nın örnekleri her iki ana grup içerisinde karışık olarak gruplanmıştır. Phylogram ağacında oluşan ana gruplar ve alt gruplar Phlomis cinsinin Phlomis ve Phlomoides olmak üzere iki alt seksiyona ayırımını desteklememektedir. Ayrıca, oluşan ana gruplar ve alt gruplar Bentham’ın yaptığı [6] Phlomis seksiyonun Oxyphlomis (pembe–mor çiçekli ve otsu bitkiler; P. Pungens P.samia P.integrifolia P.rigida), Gymnophlomis (sarı çiçekli ve otsu; P. oppositiflora, P. armeniaca, P. nissolii, P. linearis, P. physocalyx, P. capitata, P.kurdica, P.bruguieri, P.angusstusima, P.kotshyana P.sieheana, P.sintenisii, P.lanceolata, P.syriaca, P.carica, P.brunneogaleata) ve Dendrophlomis (çalı; P. lycia, P. grandiflora, P. bourgaei, P. chimerae, P. lunariifolia, P. grandiflora, P. monocephala, P. leucophracta P. furuticosa P. russeliana P. viscosa P. amanica) olmak üzere 3 alt seksiyona ayıran gruplamasıyla uyumlu değildir. Birden fazla örneği olan Phlomis türlerinin örneklerinin phylogram ağacında genel olarak türlerine özgün bir şekilde gruplanmadıkları gözlenmiştir (Şekil 3.7). ISSR verilerinin Dice benzerlik indekslerine göre hazırlanan phylogram ağacının topolojisi incelendiğinde Phlomis melezlerinin ebeveynlerine göre yerleşimi şu kategorilerde toplanabilir: (1) Melez ebeveynleri arasında yerleşmiştir. Bu yerleşme ebeveynlerden biriyle fakat kardeş (sister) takson olmayacak şekildedir (MYD 1264 kodlu P.armeniaca x carica, MYD 1282 kodlu P.grandiflora x lycia, MYD 1336 kodlu P.capitata x linearis, MYD 1504 kodlu P.bourgaei x lycia, MYD 1524 kodlu P. 51 bourgaei x chimerae, MYD 1600 kodlu P.kurdica x capitata, MYD 1676 kodlu P.bourgaei x chimerae, MYD 1985 10-B kodlu P.kurdica x oppositiflora, MYD 195818 kodlu P.physocalyx x oppositiflora, MYD 1985 10-A kodlu kurdica x oppositiflora, MYD 1958-24 kodlu P.physocalyx x oppositiflora, MYD 1367 kodlu P.armeniaca x linearis).(2) Melez atalarından birine tabandan bağlanmıştır (MYD 1818 kodlu P.capitata x nissolii, MYD 1749 kodlu P.capitata x brunneogaleata, MYD 1744-1 kodlu P.brugueri x capitata, MYD 1529 kodlu P.leucophracta x lunariifolia, MYD 1530 kodlu P.leucophracta x lunarifolia, MYD 1541 kodlu P.lunariifolia x monocephala). (3) Melez atalarından birine kladistik olarak yakın bir şekilde yerleşmiştir (MYD 1276 kodlu P.bourgaei x grandiflora,MYD 1275-1 kodlu P.bourgaei x grandiflora, MYD 1274 kodlu P.bourgaei x grandiflora, MYD 1257 kodlu P.armeniaca x nissolii, MYD 1226 kodlu P.lunariifolia x monocephala). (4) Melez diğer melezler/taksonlarla bir arada gruplanmıştır (HD 4905 kodlu P.bourgaei x lycia, MYD 1581 kodlu P.capitata x brunneogaleata, MYD 1585 kodlu P.capitata x brunneogaleata, MYD 1568-2 kodlu P.kurdica x nissolii, MYD 1420 kodlu P.kurdica x capitata, MYD 1240 kodlu P.bourgaei x chimerae, MYD 1925-1 kodlu P.nissolii x pungens, MYD 1505 kodlu P.grandiflora x lycia, MYD 1415 kodlu P.linearis x oppositiflora, MYD 1414 kodlu P.kurdica x linearis, MYD 1382 kodlu P.capitata x nissolii, MYD 1150-1 kodlu P.kurdica x capitata, MYD 1197 kodlu P.kurdica x linearis, MYD 1199 kodlu P.kurdica x linearis, MYD 1201 kodlu P.linearis x oppositiflora, MYD 1446 kodlu P.armeniaca x nissolii, MYD 1589 kodlu P.bruguieri x brunneogaleata, MYD 1595 kodlu P.bruguieri x capitata, MYD 1694 kodlu P.capitata x nissolii, MYD 1985-25 kodlu P.kurdica x oppositiflora, MYD 2736 kodlu P.armeniaca x nissolii) (Şekil 3.8). Kodu MYD 1542 olan ve bir atası bilinmeyen P.leucophracta melezi phylogram ağacında MYD 1535-3 kodlu P.lunariifolia ile kardeş takson olarak yan yana yer alırken MYD 1541 kodlu P.lunariifloia x monocephala melezi de bunlara tabandan bağlanmıştır. Kodu 1675 olan ve bir atası bilinmeyen P.leucophracta melezi çalışmada MYD 1617-2 kodlu P.linearis ve MYD 1670 kodlu P.leucophracta nın birbiriyle kardeş takson olarak yer aldıkları ve bunlara tabandan bağlanan MYD 1671-1 kodlu P.lunariifolia ile birlikte oluşturdukları gruba tabandan bağlanmıştır (Şekil 3.9). 52 1 3 6 2 7 9 8 12 15 17 18 16 21 23 22 13 14 19 20 4 5 11 10 24 26 25 28 29 27 30 31 35 32 33 34 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 53 52 54 55 56 60 58 59 62 63 64 66 67 65 100MW 0.30 0.46 0.62 0.78 Coefficient Şekil 3.7. Phlomis örneklerinde ISSR verileri ile oluşturulan phylogram ağacı parça 1 0.94 53 68 57 61 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 90 91 89 115 116 117 119 120 121 118 126 122 123 124 125 127 131 130 132 133 134 137 135 136 128 129 138 144 148 139 140 141 145 146 147 149 154 150 151 152 153 155 100MW 0.30 0.46 0.62 0.78 Coefficient Şekil 3.8. Phlomis örneklerinde ISSR verileri ile oluşturulan phylogram ağacı parça 2 0.94 54 156 157 158 159 142 143 92 93 94 95 96 97 99 100 101 102 104 103 105 106 107 108 109 112 113 110 111 114 183 184 185 189 186 187 188 190 191 192 193 98 174 175 176 177 178 179 180 181 182 160 171 172 173 161 162 163 169 170 164 165 166 167 168 100MW 0.30 0.46 0.62 0.78 Coefficient Şekil 3.9. Phlomis örneklerinde ISSR verileri ile oluşturulan phylogram ağacı parça 3 0.94 55 3.2.2 ISSR verilerine göre hazırlanan Network analizi ISSR verilerinin network analizi yapılırken; Dice benzerlik indeksleri 1’den çıkarılarak genetik mesafeler hesaplanmıştır. Network analizleri de genetik mesafeler üzerine kurulmuştur. Network oluşturulurken Splits Tree 4 (versiyon 4,13,1) programı kullanılmıştır. ISSR verilerinin neigbour – network modeline uyumu yüksek olup %99,69 ‘dur (fit=95.58, LSfit= 99.69). Network grafiğinin topolojisi incelendiğinde network ün merkezinde bir nedeni retikülat olaylar da olan verideki uyumsuzlukları gösteren çok sayıdaki paralel kenarlardan oluşan kutucuklar yer almaktadır. Benzer şekilde, network grafiğinde oluşan grupların içerisinde de çok sayıdaki paralel kenarlardan oluşan kutucuklar yer almaktadır. Bu bulgular araştırmada kullanılan Phlomis örneklerinin uzak ve yakın tarihsel geçmişinde melezleşme gibi retikülat olayların yer aldığını göstermektedir. Network grafiği incelendiğinde 46 adet melez ve 36 farklı taksondan oluşan, 193 farklı Phlomis örneğinin 10 farklı grup oluşturarak dağıldığı fakat bu grupların Phlomis alt seksiyonlarına uyumlu olarak dağılmadığı görülmektedir. Gruplar incelendiğinde birden fazla örneği olan Phlomis türlerinin örneklerinin genel olarak türlerine özgün bir şekilde bir arada toplanmadığı görülmektedir. Gruplara genel olarak melezlerin yerleşimi açısından bakıldığında melezlerin ya atalarından biriyle yan yana yer aldığı veya atalarından birinin veya her ikisinin de bulunduğu grupta yer aldığı görülmektedir (Şekil 3.10 ve 3.11). Fakat az sayıda melezin atalarından birisinin veya her ikisinin de bulunmadığı bir grupta yer aldığı da görülmektedir. ISSR verileri kullanılarak Dice benzerlik indeksine göre elde edilmiş benzerlik değerlerinin birden çıkartılmasıyla elde edilen genetik mesafe matriksi üzerine kurulan Neighbour-net ağ (network) analizinde Phlomis melezlerinin ebeveyinlerine göre yerleşimleri şu kategorilerde toplanabilir: (1) Melez atalarından birinin hemen yanında yer almaktadır (MYD 1264 kodlu P. armeniaca x carica, MYD 1367 kodlu P. armeniaca x linearis, MYD 1420 kodlu P. capitata x kurdica, MYD 1676 kodlu P. bourgaei x chimerae, MYD 1568-2 kodlu P. kurdica x nissolii, MYD 1600 kodlu P. kurdica x capitata, MYD 1541 kodlu P. lunariifolia x monocephala, MYD 1749 kodlu P. capitata x brunneogalata, MYD 1818 kodlu P. capitata x nissolii, MYD 1958-24 kodlu P. physocalyx x oppositiflora, MYD 1985-10-A P. kurdica x oppositiflora, 56 MYD 1958-18 kodlu P. physocalyx x oppositiflora, MYD 1985-10B kodlu P. kurdica x oppositiflora, MYD 1524 kodlu P. bourgaei x chimerae, MYD 1530 kodlu P. leucophracta x lunariifolia, MYD 1529 kodlu P. leucophracta x lunariifolia, MYD 1504 kodlu P. bourgaei x lycia, MYD 1694 kodlu P. capitata x nissolii, MYD 1744-1 kodlu P. bruguieri x capitata) (2) Melez atalarının birisinin veya her ikisinin bulunduğu grupta yer almaktadır (MYD 1226 kodlu P. lunariifolia x monocephala, MYD 1257 kodlu P. armeniaca x nissolii, MYD 1240 kodlu P. bourgaei x chimarea, MYD 1274 kodlu P. bourgaei x grandiflora, MYD 1275-1 kodlu P. bourgaei x grandiflora, MYD 1276 kodlu P. bourgaei x grandiflora, MYD 1282 kodlu P. grandiflora x lycia, MYD 1336 kodlu P. capitata x linearis, MYD 1415 kodlu P. linearis x oppositiflora, MYD 1414 kodlu P. kurdica x linearis, MYD 1382 kodlu P. capitata x nissolii, MYD 1585 kodlu P. capitata x brunneogalata, MYD 1581 kodlu P. capitata x brunneogalata, 1595 kodlu P. bruguieri x capitata, MYD 1985-25 kodlu P. kurdica x oppositiflora, MYD 1446 kodlu P. armeniaca x nissolii, MYD 1505 kodlu P. grandiflora x lycia, MYD 1925-1 kodlu P. nissolii x pungens). (3) Melez atalardan birinin veya ikisinin de yer almadığı grupta bulunmaktadır (MYD 1197 kodlu P. kurdica x linearis, MYD 1199 kodlu P. kurdica x linearis, MYD 1201 kodlu P. linearis x oppositiflora, MYD 1589 kodlu P. bruguieri x brunneogalata, HD 4905 kodlu P. bourgaei x lycia, MYD 2736 kodlu P. armeniaca x nissolii) (Şekil 3.10 ve 3.11 ). Bir atası belirlenmemiş olan ve diğer atasının P.leucophracta olduğu bilinen MYD 1675 ve 1542 kodlu P.leucophracta melezleri network grafiğinde incelendiğinde MYD 1542 P.leucophracta melezi, MYD 1541 kodlu P.leucophracta x monocephala ile 1535-3 kodlu P.lunariifolia arasında yer almıştır. MYD 1675 kodlu P.leucophracta melezi ise MYD 1670 P.leucophracta’ nın hemen bir yanında yer almış ve MYD 1670 P.leucophracta’ nın diğer yanında ise MYD 1671-1 kodlu P.lunariifolia yer almıştır. Bu bulgular MYD 1675 ve 1542 kodlu P.leucophracta melezlerinin ikinci atasının P.lunariifolia olduğu yönünde güçlü destek sunmaktadır (Şekil 3.10 ve 3.11 ). 57 Şekil 3.10. Phlomis örneklerinde ISSR verilerine ait neighbour- network analizi (1. parça) 58 Şekil 3.11. Phlomis örneklerinde ISSR verilerine ait neighbour- network analizi (2. parça) 59 4. BÖLÜM TARTIŞMA VE SONUÇ 4.1. TARTIŞMA Ülkemizde Lamiaceae familyası 45 cins, 565 tür ve toplam 765 taksonla temsil edilmektedir. Endemizm oranı % 45 olarak tespit edilmiştir [4]. Lamiaceae familyasının içerisinde yer alan Phlomis cinsi Dadandı (2002)’nın “Türkiye’nin Phlomis L. (Lamiaceae) Cinsi Revizyonu” adlı doktora tezinde 39 tür ve tür altı, ve 19 melezle toplamda ise 58 takson ile sınıflandırılmıştır. Phlomis cinsinin endemizm oranı % 58,8 gibi yüksek bir orana sahiptir [3]. Bu durum Anadolu’nun gen merkezlerinden biri olmasından kaynaklanmaktadır. Phlomis cinsinin 19 meleze sahip olması bu cinste melezleşmenin oldukça yüksek olduğunun göstergesidir. Yüzbaşıoğlu ve arkadaşlarının yapmış olduğu çalışma sonucunda P. x termessi’nin morfolojik ve moleküler olarak atalarının arasında yer aldığı gözlenmiştir [22]. Çalışmada görülmüştür ki gerek morfolojik gerekse moleküler olarak melezler ata türler arasında bir mozaik oluşturmuştur. Bu çalışmada Phlomis cinsine ait 46 adet melezin ataları ile ilişkisi 24 farklı karakterde morfolojik olarak ve ISSR metodu ile moleküler olarak incelenmiş ve görülmüştür ki bazı melezler atalarının ikisi arasında yer almış ya da en az birine bağlanmıştır. Bazı melez bireyler de atalarından farklı olarak diğer tür ya da melezler ile bağlanmıştır. Phlomis seksiyonu, sarı çiçekli ve otsu Gymnophlomis alt seksiyonu, odunsu Dendrophlomis alt seksiyonu, pembe–mor çiçekli ve otsu bitkilerden oluşan Oxyphlomis alt seksiyonu olmak üzere 3 alt seksiyona ayrılmaktadır [6]. Yapılan bu araştırmada gerek morfolojik gerekse moleküler verilerden elde edilen phylogenetik ağaçlarda bu ayrıma göre bir dağılma olmadığı görülmektedir. Hem morfolojik veriler 60 hem de ISSR verileri kullanılarak hazırlanan Phylogram ağaçlarının her birinde Phlomis taksonları ve melezlerinin 2 ana gurupta toplandığı fakat bu dağılımın Phlomis alt seksiyonlarına göre olmadığı görülmüştür (Şekil 3.1,3.2,3.3). Morfolojik verilere göre oluşturulan ağaçta melezlerin bazılarının bir atasına kladistik olarak yakın yer aldığı bazılarının ise atalarından birisiyle kardeş takson olduğu ama melezlerin çoğınluğunun ise atalarından uzakta yer aldığı ve diğer türler ve melezlerle gruplandığı görülmüştür. MYD 1199 kodlu P.kurdica x linearis; MYD 1367 kodlu P. armeniaca x linearis; MYD 1420 kodlu P. kurdica x capitata; MYD 1150-A-1 kodlu P. kurdica x capitata; MYD 1226 kodlu P. lunariifolia x monocephala; MYD 1264 kodlu P. armeniaca x carica; MYD 1446 kodlu P. armeniaca x nissolii; MYD 1524 kodlu P. bourgaei x chimerae; MYD 1541 kodlu P. lunariifolia x monocephala; MYD 1568-2 kodlu P. kurdica x nissolii; MYD 1585 kodlu P. capitata x brunneogaleata; MYD 1581 kodlu P. capitata x brunneogaleata; MYD 1676 kodlu P.bourgaei x chimerae; MYD 1694 kodlu P. capitata x nissolii; MYD 1925-1 kodlu P. nissloii x pungens; MYD 1197 kodlu P. kurdica x linearis; MYD 1240 kodlu P. bourgaei x chimerae; MYD 1257 kodlu P. armeniaca x nissolii; MYD 1274 kodlu P. bourgaei x grandiflora; MYD 1282 kodlu P. grandiflora x lycia; MYD 1336 kodlu P.capitata x linearis; MYD 1382 kodlu P.capitata x nissolii; MYD 1414 kodlu P.kurdica x linearis; MYD 1505 kodlu P.grandiflora x lycia; MYD 1595 kodlu P.bruguieri x capitata; MYD 1600 kodlu P.kurdica x capitata; MYD 1744-1 kodlu P.bruguieri x capitata; MYD 1749 kodlu P.capitata x brunneogaleata; MYD 1985-10-B kodlu P.kurdica x oppositiflora; MYD 1958-18 kodlu P.physocalyx x oppositiflora; MYD 1985-10-A kodlu P.kurdica x oppositiflora; MYD 1958-24 kodlu P.physocalyx x oppositiflora; HD 4905 kodlu P.bourgaei x lycia; MYD 1542 kodlu P.leucophracta melezi; MYD 1589 kodlu P.bruguieri x brunneogaleata; MYD 1585 kodlu P.capitata x brunneogaleata; MYD 1675 kodlu P. leucophracta melezi melezleri atalarından farklı gruplanmıştır (Şekil 3.1, 3.2, 3.3 ). Morfolojik verilere ait neighbour- network analizinde Phlomis tür ve melezlerinin genel olarak 2 ana grupta toplandığı görülmüş ve birinci grupta genellikle Gymnophlomis alt seksiyonuna ait örneklerin yer aldığı ikinci grupta ise genellikle Dendrophlomis alt seksiyonuna ait türlerin yer aldığı görülmüştür. Oxyphlomis alt seksiyonu örnekleri ise 2 grupta da bulunmuştur (Şekil 3.4 ve 3.5). 61 P.kurdica x linearis MYD 1197 ve P.bruguieri x capitata MYD 1595 melezleri hem morfolojik hem de moleküler phylogram ağaçlarında atalarından bağımsız dallanmıştır ( Şekil 3.1, 3.2, 3.3 ve 3.7, 3.8, 3.9). ISSR verilerine ait neighbour- network analizi incelendiğinde melezlerin ataları ile aralarında ilişki oldukça açık görülmektedir. Melezler ataları ile genellikle bir veya birkaç kutucuk, paralel ile bağlandığı bazılarının atalarının arasında yer aldığı görülmüştür. Araştırmada kullanılan melezlerin 9 tanesinin atalarından birinin hemen yanında yer almış olduğu, 28 tanesinin atalarının birisinin veya her ikisinin bulunduğu grupta yer almış olduğu ve 9 tanesinin ise atalarından ikisininde bulunmadığı grupta yer almış olduğu gözlenmiştir. MYD 1529 ve 1530 kodlu P.leucophracta x lunarifolia melezlerinin ataları aynı olmakla birlikte MYD 1526 P.lunairifolia ve MYD 1527 P.leucophracta atalar birbiri ile aynı kutucuğun farklı kenarlarına bağlanmıştır. Melez de ataların kutucuğuna bir kutucuk ile bağlanarak yanlarında yer almıştır (Şekil 3.10 ve 3.11 ). Bir atası bilinmeyen MYD 1542 kodlu P.leucophracta melezi ISSR verileri ile oluşturulan filogenetik ağaçta MYD 1535-3 kodlu P.lunariifolia ile kardeş takson olarak yan yana yer alırken MYD 1541 kodlu P.lunariifloia x monocephala melezi de bunlara tabandan bağlanmıştır. Morfolojik verilere ait phylogenetik ağaçta ise MYD 1585 P.capitata x brunneogaleata ve MYD 1589 P.bruguieri x brunneogaleata ile aynı dal üzerinde bir grup oluşturmuştur (Şekil 3.1, 3.2 ve 3.3 ). Morfolojik veriler ile düzenlenen network analizinde ise Gymnophlomis alt seksiyonuna ait türler arasında yer almıştır (Şekil 3.4 ve 3.5 ). ISSR verilerine göre düzenlenmiş neighbour- network analizinde ise phylogram ağacına paralellik göstererek MYD 1541 kodlu P.leucophracta x monocephala ile MYD 1535-3 kodlu P.lunariifolia arasında yer almıştır. (Şekil 3.10 ve 3.11 ). Bir atası bilinmeyen diğer melez örneği MYD 1675 P.leucophracta melezi ISSR sonuçları ile yapılmış phylogenetik ağaçta MYD 1617-2 P.linearis, MYD 1670 P.leucophracta ve MYD 1671-1 P.lunariifolia ile bir gurup oluşturarak yan yana yer almıştır (Şekil 3.7, 3.8 ve 3.9). Morfolojik verilere ait ağaçta ise MYD 1179-8 P.amanica ile aynı dala bağlanarak, MYD 1186 P.viscosa ile aynı grupta yer alarak 62 tabandan bağlanmıştır (Şekil 3.1, 3.2 ve 3.3 ). ISSR verilerine ait neighbour-network analizinde moleküler ağaca paralel olarak MYD 1670 P.leucophracta’ nın hemen bir yanında yer almıştır (Şekil 3.10 ve 3.11). Morfolojik veriler ile oluşturulan network analizinde ise Dendrophlomis alt seksiyonu üyeleri arasında yer almıştır. ISSR verileri üzerine kurulu olan filogenetik ağaçtan ve network grafiğinden elde edilen bu bulgular MYD 1675 ve 1542 kodlu P. leucophracta melezlerinin ikinci atasının P.lunarifolia olabileceği yönünde güçlü destek sunmaktadır Filogenetik ağaçlar, genellikle melezleşme gibi evrimsel olayları açıklığa kavuşturmakta yetersiz kalırlar ve tutarsız sonuçlar gösterebilirler. Bu sebeple çalışmada network analizi de yapılmıştır. Network analizleri morfolojik verilere göre hazırlanan filogenetik ağaçtan kısmen farklılık gösterse de genel olarak benzer sonuçlar ortaya çıkarmıştır. Bu çalışmada 46 adet melez 36 farklı taksondan oluşan toplam 193 Phlomis örneğinin morfolojik açıdan bakıldığında Manhattan mesafeleri kullanılarak oluşturulan filogenetik ağaçta tüm tür ve melezlerde bir gruplaşma görülmediği ve değişkenlik gösterdiği açıkça görülürken morfolojik network analizinde bunun tam tersine alt seksiyonlarına göre ayrılan aynı türlerin bir arada yer aldığı görülmüştür. Melezlerin ataları ile olan ilişkisi incelendiğinde hem filogenetik ağaçta hem de network grafiğinde bazı melezler, atalardan en az birine bağlanırken bazıları atası olmayan bir tür ya da başka bir melezle bir arada yer almıştır. Örneğin MYD 1415 kodlu P. linearis x oppositiflora melezi atası 1961 P.oppositflora ile bağlanırken, 1985-25 P.kurdica x oppositiflora melezi atası olmayan 1932-7 P.nissolii ile bağlanmıştır (Şekil 3.2). Evren Ö.H. (2012) yüksek lisans tezinde Phlomis kurdıca ve Phlomis opposıtıflora türlerinde tür içi genetik varyasyonu araştırmış ve Phlomis x melitenense’ nin (Lamiaceae) melezliğini moleküler yönden incelemiş ve çalışmasında hem ISSR hem de RAPD markerlarını kullanmıştır. Araştırmada kullanılan ISSR primerlerinden elde edilen bant sayısı 651 olup, bu bantlardan yalnız 1 tanesi monomorfiktir [26]. Benzer bir şekilde Wright (2007) Hawai’de üç ISSR primeri ile 287 Metrosideros polymorpha Gaudich. türünü genetik olarak incelemiş, 111 bant profili elde etmiş ve elde ettiği 111 bantın da polimorfik olduğunu belirtmiştir [72]. 63 Bu çalışmada ISSR marker’ları ile oluşturulan bant profiline bakıldığında toplam bant sayısının 451 ve bantların %100 polimorfik olduğu görülmüştür. Hiç monomorfik banda rastlanmamıştır ve bu açıdan elde edilen bulgular önceki iki aratırmanın sonuçlarıyla paralellik göstermektedir. Öztoprak F. (2015) yüksek lisans tezinde kloroplast DNA analizi ile Phlomis L. (Lamiaceae) cinsine ait türler arasındaki doğal hibridizasyonu incelemiş ve çalışmasında benzer olarak filogram ağacı ve neighbour- network analizine yer vermiştir. Çalışma sonuçları büyük ölçüde paralellik gösterirken filogenetik ağaçlar ve network analizleri büyük ölçüde birbirini desteklemiştir [28]. 4.2 SONUÇ Sonuç olarak Phlomis cinsine ait 46 adet melez ve 36 farklı taksonda melezleşme ve genetik ilşki ISSR yöntemi ve morfolojik özellikler bakımından incelenmiş olup, elde edilen morfolojik ve moleküler filogenetik ağaçlarda ve yine morfolojik ve moleküler filogenetik networklerde örneklerin alt seksiyonlara uygun bir gruplanma göstermediği görülmüştür. Network analizlerinde melez örneklerin büyük bir kısmının atası kabul edilen türlerin hemen yanında veya onların bulunduğu grupta yer aldıkları gözlenirken bir kısmının ise başka türler ve melezler ile bir arada gruplandıkları gözlenmiştir. Network analizleri araştırmada kullanılan Phlomis örneklerinin evrimsel tarihinde retikülat olayların olduğuna işaret etmektedir. Phlomis taksonlarının gruplanması bakımından filogenetik ağaç ve filogenetik network analizleri benzerlik göstermekte ve morfolojik ve moleküler sonuçlar da büyük oranda paralellik göstermektedir. Melezleşmenin açıklanmasında filogenetik network filogenetik ağaca göre daha başarılı sonuçlar vermiştir. Sonuçlar bir atası bilinmeyen 1675 ve 1542 kodlu P. leucophracta melezinin hem filogenetik hem de network analizinde 1535-3 P. lunarifolia türü ile yan yana yer aldığı, ISSR verilerinin network analizinde bu tür ile aynı kutucuk üzerinde yer alarak diğer atası olabileceğini düşündürmektedir. 64 KAYNAKLAR 1. Azizian, D., Moore, D. M., 1982. Morphological and palynologial studies in Phlomis L.,Erumostachys Bunge and Paraphlomis Prain (Labiateae). Botanical Journal of Linnean Society, Vol. 185: 225-248. 2. Huber-Morath, A.,1982. Phlomis L. In Flora of Turkey and East Aegean Islands, Davis, P. H. (Ed), Edingburg Univ. Press., Vol.7, Edinburg. 1982, Pp. 102-126. 3.Dadandı, M. Y., 2002. Türkiye’nin Phlomis L. (Lamiaceae) Cinsi Revizyonu, Doktora Tezi, Gazi Üniversitesi, Ankara. 4. Davis, P.H., Mill, R.R., Tan, K., 1988. Flora of Turkey and East Aegean Islands Supplement I, Davis P. H.(Ed) Edinburg Univ. Press., Vol. 10, Edinburg, Pp; 474-505. 5 .Moench, C., 1794. Methods Plantas Horti Botanici et Agri Marburgensis a Stramium Situ Describendi. Marburgi Cattorum, Marburg, Germany. 6. Bentham, G., 1834. Labiatarum Genera et species, London, p:323-644. 7. Baytop. T., 1994. Türkçe Bitki Adları Sözlüğü, Türk Dil Kurumu Yayınevi 578, Ankara. 8. Coombes, A. J., 2002. Plant Names The A to the Z, Timber Press, USA,Pp. 312. 9. Baytop. T., 1984. Türkiye’de Bitkilerle Tedavi, İst. Üniv. Yay., No. 3255, İstanbul. 10. Baytop, T., 1999. Therapy with Medicinal Plants in Turkey, Past and Present, 2nd ed, Nobel Tıp Basımevi, İstanbul, Turkey, ISBN: 9754200211, 193 p. 11. Gürbüz, I., Üstün O., Yeşilada, E., Sezik, E., Kutsal, O., 2003. Anti-ulcerogenic activity of some plants used as folk remedy in Turkey, Journal of Ethnopharmacolgy, Vol 88, no: 1: 93-97. 12. Seçmen, Ö., Gemici, Y., Görk, G., Behçet, L., Leblebici, E., 2004. Tohumlu Bitkiler Sistematiği, Ege Üniversitesi Basımevi, İzmir, 582.1, s : 276. 13. Hedge, I.C., 1986. Labiatae of south-west Asia: diversity, distribution and endemism. Proceedings of the Royal Society of Edinburg, Vol. 89B, 23-35. 14. Arnold, M. L., 1997. Natural Hybridisation and Evolution. Oxford Universty Press, New York, New York, USA. 65 15.Rieseberg, L. H., Carney, S. E., 1998.Tansley Review No. 102 Plant Hybridization, New Phytologist. 140, 599-624. 16.Burke, J.M., 2000. Hybrids and Hybridization, Plant Sciences for Students, Macmillan, USA, New York. Pp: 32-35. 17. Paun, O., Forest, F., Fay, M. T., Chase, M. W., 2009. Hybrid speciation in angiosperms: Parental divergence drives ploidy, New Phytologist, 182: 507518. 18. Seehausen, O., 2004. Hybridization and adaptive radiation. Trends in Ecology and Evolution, 19(4): 197-207. 19. Whitham, T. G., Martinsen, G. D., Floate, K. D., Dungey, H. S., Potts, B. M., Keim, P., 1999. Plant hybrid zones affect biodiversty; tools for a genetic-based understanding of community structure. Ecology 80: 416-428. 20. Albaladejo, R.G., A guilar, J.F., Aparicio, A., Feliner, N.G., 2005. Contrasting nuclear-plastidial phylogenetic patterns in the recently diverged Iberian Phlomis crinita and P. lychnitis lineaces (Lamiaceae), Taxon, 54(4):987-998. 21. Pan, Y.Z., Fang, L.Q., Hao, G., Cai, J., Gong, X., 2009. Systematic positions of Lamiophlomis and Paraphlomis (Lamiaceae) based on nuclear and chloroplast sequences, Journal of Systematics and Evolution, 47, 535-542 22. Yüzbaşıoğlu, E., Dadandı, M. Y., 2008. Phylogenetic relationship among species of the subsection Dendrophlomis Bentham, Electronic Journal of Biotechnology, Vol.11, No:4, 1-9. 23. Yüzbaşıoğlu, E., Dadandı, M.Y., Özcan, S., 2008. Natural hybridization between P. lycia D. Don x P. bourgaei Boiss., (Lamiaceae) revealed RAPD markers, Genetica, 133, 13-20. 24. Dadandı, M.Y., Yüzbaşıoğlu, E., Duman, H., Özcan, S., P. x termesi P.H. Davis (P. bourgaei Boiss. x lycia D. Don) Taksonunun Morfolojik ve Moleküler Yöntemlerle İncelenmesi, XVII. Ulusal Biyoloji Kongresi, 21-24 Haziran 2004, Adana, 5. Seksiyon, pp.94 66 25. Yüzbaşıoğlu, E., Dadandı, M. Y., Özcan, S., 2009. Estimation of phylogenetic relationship of Phlomis species based on seed protein polymorphism. Electronic Journal of Biotechnology, ISSN: 0717-3458, Vol. 12 No. 2. Pp. 1-9. 26. Evren, Ö. H., 2012. Phlomis kurdica ve P. oppositiflora Türlerinde Tür İçi Genetik Varyasyonun Belirlenmesi ve P. x melitenense’nin Melezliğinin Moleküler Yönden İncelenmesi. Akdeniz Üniversitesi, Yüksek Lisans Tezi, Antalya, 191 s. 27. Sezer, G., 2012. Phlomis physocalyx ve P. oppositiflora Türlerinde Tür İçi Genetik Varyasyonun Moleküler Düzeyde Belirlenmesi. Erciyes Üniversitesi, Yüksek Lisans Tezi, Kayseri, 243 s. 28. Öztoprak, F., 2015. Kloroplast DNA Analizi ile Phlomis L. (Lamiaceae) Cinsine Ait Türler Arasındaki Doğal Hibridizasyonun İncelenmesi. Erciyes Üniversitesi, Yüksek Lisans Tezi, Kayseri, 110 s. 29. Reddy, M. P., Sarla, N., Siddiq, E. A., 2002. Inter simple sequence repeat (ISSR) polymorphism and its application in plant breeding. Euphytica 128: 9-17. 30. Wang, G., Mahalingan, R., Knap, H. T., 1998. (C-A) and (G-A) anchored simple sequence repeats (ASSRs) generated polymorphism in soybean, Glycine max (L.) Merr. Theoretical and Applied Genetics, 96: 1086-1096 31. Wang, H. Z., Wu, Z. X., Lu, J. J., Shi, N. N., Zhao, Y., Zhang, Z. T., Liu, J. J., 2008. Molecular diversity and relationships among Cymbidium goeringii cultivars based on inter-simple sequence repeat (ISSR) markers. Genetica 136(3): 391399. 32. Carrodus, S. K., 2009. Identification and the Role of Hybridisation in New Zealand Pittosporum, Yüksek Lisans Tezi, The Universty of Waikato, Yeni Zelanda, 150 p. 33. Metcalfe, C.R., Chalk., L., 1950. Anatomy of the Dicotyledones. Oxford Univesrty Press, Londra, 2, 1041 pp. 34. Güner, A., Aslan, S., Ekim, T., Vural, M., Babaç, M. T. (edlr), 2012. Türkiye Bitkileri Listesi (Damarlı Bitkiler), Nezehat Gökyiğit Botanik Bahçesi ve Flora Araştırmaları Derneği Yayını, İstanbul. 67 35. Ekim, T., Koyuncu, M., Vural, M., Duman, H., Aytaç, Z., Adıgüzel, N., 2000. Red Data Book of Turkish Plants (Pteridophyta and Spermatophyta). TTKD and Van Centennian Universty Press, Ankara, Turkey, ISBN: 975-93611-0-8: 100-171. 36. Taylor, J. M., 1998. Phlomis, the Neglected Genus, a Guide for Gardeners and Horticulturists.National Council for the Conservation of Plants and Gardens, Colouster, England. ISBN: 0953241300, Pp. 387. 37. Aparicio, A., Albaladejo, R. G., Porras, M., Ceballos, G., 2000. Isozyme evidence for natural hybridisation in Phlomis (Lamiaceae): Hybrid origin of the Rare P. x margaritae, Annals of Botany, 85: 7-12. 38. Demirci, B., Başer, K. H., Dadandı, M. Y., 2004. Composition of the Essential Oils Phlomis rigida Labill. And P. samia L. 35th International Symposium on Essential Oils (USEO2004), Pp.133, September29-October2, Messina, Italy. 39. McDade, L. A., 1992. Hybrids and phylogenetic systematics II: The impact of hybrids on cladistic analyses, Evolution, (46)5: 1329-1346. 40. Karcıcıo, M., 2006. Analysis of Genetic Diversity in Landraces of Macoroni Wheat (Triticum durum Desf.) by RAPD Technique, Doktora Tezi, Hacettepe Üniversitesi, Ankara. 41. Moritz, C., Hillis, D.M., 1990. Molecular Systematics: Context and Controversies, Sinauer Associates, Inc. Sunderland, Massachusetts, USA, Pp. 1-16. 42. Williams, J. G. K., Kubelik, A. R., Livak, K. J., Rafalski, J. A., Tingey S. V., 1990. DNA polymorphism amplified by arbitrary primers are useful as genetic markers, Nucleic Acids Research 18: 6231-6235. 43. Özcan, S., Gürel, E., Bababoğlu, M., 2004. Bitki Biyoteknolojisi II Genetik Mühendisliği ve Uygulamaları, Selçuk Üniversitesi, Konya. 44. Khan, S., Spoor, W., 2001. Use of molecular and morphologic markers as a quality control in plant tissue culture. Pakistan Journal of Biological Sciences 4/4: 479-482. 45. Bretting, P. K., Widrlechner, M. P., 1995. Genetic markers and horticultural germplasm management, Hortscience, 30(7), 1329 – 1356. 68 46. Weising, K., Nybom, H., et al., Wolffe, K., Meyer, W., 1995. DNA fingerprinting in plants and fungi, CRC Press, Pp. 12 – 13, Boca Raton, FL. 47. Staub, J. E., Serquen, F. C., Gupta, M., 1996. Genetic markers map construction and their application in plant breeding. Hortiscience 31: 729-741. 48.Kephart, S. R., 1990. Starch electrophoresis of plant isozymes, a comparative analysis of thecniques, American Journal of Botany, 77, 693-712. 49. Tanksley, S. D., 1983. Molecular markers in plant breeding. Plant Molecular Biology Reporter, 11, 3-8. 50. Joshi, S. P., Gupta, V. S., Aggarwal R. K., Ranjekar, P. K., Brar, D. S., 2000. Genetic diversity and phylogenetic relationship as revealed by inter simple sequence repeat (ISSR) polymorphism in the genus Oryza,Theoretical and Applied Genetics, 100: 1311-1320. 51. Wolf, A. N. D., Liston, A., 1998. Contributions of PCR Based Methods to Plant Systematics, Molecular Systematics of Plants II. Chopman and Hall, New York, 33-86 52. Kesawat, M. S., Das, B., K., 2009. Molecular Markers: It’s application in crop improvement, Journal of Crop Science and Biotechnology 12(4):169-181. 53. Filiz. E., Koç. İ., 2011. Bitki biyoteknolojisinde moleküler markörler, GOÜ, Ziraat Fakültesi Dergisi, 28(2), 207-214. 54. XU, D. H.., Kanazawa, J. A.A., Gai, J.Y., and Shimamoto, Y., 2001. Identification sequence variations by PCR-RFLP, and its application to the evolution of cpDNA diversty in wild and cultivated soybeans,Theoretical and Applied Genetics, 102(5): 683-688. 55. Gupta, P. K., Chyi, Y.-S.,Romero-Severson, J., Owen, J. L., 1994. Amplification of DNA markers from evolutionary diverse genomes using simple primers of simple sequence repeats, Theoretical and Applied Genetics, 89, 998-106. 56. Ergül, A., 2000. Asmalarda (Vitis vinifera L. Cvs) Genomik DNA Parmakizi Analizleri ile Moleküler Karakterizasyon, Doktora Tezi. Fen Bilimleri Enstitüsü, Bahçe Bitkileri Anabilim Dalı, Ankara Üniversitesi, Ankara. 69 57. Vos, P., Hogers, R., Bleeker M., 1995. AFLP a new technique for DNA fingerprint, Nucleic Acids Research, 23: 4407-4419. 58. Walton M., 1993. Molecular markers: Which ones to use?,Seed World, July, 23 – 29 59. Backeljau, T., Bruyn, L. D., Wolf, H. D., Jordaens, K., Dongen, S. V., Verhagen, R., Winnepenninckx, B., 1995. Random amplified polymorphic DNA (RAPD) and parsimony methods, Cladistic, 11, 119-130. 60. Paran, I., Michelmere, R.W., 1993. Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce, Theorotical and Applied Genetics, 85, 985-993. 61. Alvarez, I., Wendel, J. F., 2003. Nuclear ribosomal spacer regions in plant phylogenetic inference, Molecular Phylogenetics and Evolution, 29, 417-434. 62. Poczai, P., Hyvönen, J., 2010. Nuclear ribosomal spacer regions in plant phylogenetics: Problems and prospects, Molecular Biology Reporter, 37, 18971912. 63. Baldwin, B. G., Sanderson, M. J., Porter, J . M., 1995. The ITS region of nuclear ribosomal DNA: a valuable source of evidence on angiosperm phylogeny, Annals of the Missouri Botanical Garden 82: 247-277. 64. Poczai, P., Hyvönen, J., 2010. Nuclear ribosomal spacer regions in plant phylogenetics: Problems and prospects, Molecular Biology Reporter, 37, 18971912. 65. Gupta, P. K., Chyi, Y.-S.,Romero-Severson, J., Owen, J. L., 1994. Amplification of DNA markers from evolutionary diverse genomes using simple primers of simple sequence repeats, Theoretical Applied Genetics, 89, 998-106. 66. Karslı, T., Karabağ, K., vd. DNA Marker Yöntemleri ve Hayvancılıkta Kullanımı, 100. Yıl Üniversitesi, Van, II. Ulusal Zootekni Öğrenci Kongresi, Mayıs 2006. 67. Godwin ID, Aitken AB, Smith L. W., 1997. Application of inter simple sequnece repeat (ISSR) markers to plant genetics, Electrophoresis, 18, 1524-1528. 70 68. Parker, L., Bordolla, P., Colova, V., 2005. Tracing the pedigree of cynthiana grape by DNA microsatellite markers. Proceedings of the Florida State Horticultural Society 118: 200-204. 69. Moreno, S., Martian J. P., Ortiz, J. M., 1998. Inter-simple sequence repeats PCR for characterization of closely related grapevine germplasm, Euphytica 101: 117125. 70. F. James Rohlf, NTSYSpc Numerical Taxonomy and Multivariate Analysis System Version 2.2 2009 by Applied Biostatistics Inc., 10 Inwood Road, Port Jefferson, New York 11777. 71. Huson, D. H., Bryant, D., 2006. Application of phylogenetic networks in evolutionary studies, Molecular Biology and Evolution, 23(2):254-267. 72. Wright, M. E., 2007, B.S. Brigham Young University, Thesis of Master, USA 71 EKLER EK 1. Phlomis örneklerinde incelenen morfolojik karakterlere ait ölçüm değerleri Örnek no. A B C D G H I J 1 2 6,2 16 16 16 6 2,1 2,5 2 2 4 14 15 16 4 1,5 2 3 3 7 24 24 24 6,5 4 2,5 4 4 7,5 30 30 30 5 5 6 26 26 26 6 3 4 10 10 10 7 3 5,5 24 24 24 8 3 6,5 10 10 10 9 3 5 10 4 4,5 8 11 3 2,5 24 24 24 12 2 8 24 24 24 13 2 2,5 8 8 14 3 5,5 12 12 12 15 2 8,7 24 24 24 16 2 3,5 6 6 17 2 5 18 18 18 7 2 2,5 18 1 18 18 18 6 2,2 19 5 2,3 6 6 6 12 3,9 20 3 3 6 6 6 21 3 3 8 8 8 22 8 5,5 15 15 15 23 4 3 8 8 8 4 1 2 5 24 3 8,5 6 6 6 21 15 20 13 25 3 3 6 7 8 11 2,5 8 26 3 5 6 7 8 27 1 28 4 29 6 30 1 31 2 32 8 8 6 8 K Q R S T U V W X Y Z 1 0,5 0,5 1,5 2,6 2 2 2 1 2 1 0,5 0,2 1,2 0,5 2 2 2 1 2 3 1,7 0,6 1,2 2,2 3,2 2 2 2 1 3 4 1,2 1,1 0,3 0,4 0,8 1,5 1 1 1 1 1 5 3 1 0,3 0,2 1 1,8 1 1 1 1 1 10 1,3 1,3 0,5 0,2 0,6 2 2 2 1 1 1 14 7 1,6 1,5 0,6 0,5 2,5 4 2 2 1 1 1 5,5 7 0,4 1 0,7 0,4 1 2,5 2 2 1 2 2 1,5 0,6 2,4 3,5 2 2 1 1 1 2,2 2 2 1 2 2 5 L 2 14 14 7 7,7 7,5 0,7 1,5 13 8 4,5 13 2,4 10 N O 2 5,2 2,6 0,5 3 1,5 7 2,5 4 3 1 1 0,5 9 3 9 8 7,5 1 99 4,5 1,1 0,7 0,4 0,4 1 3 3 1,3 0,9 0,4 0,3 1 1 1 1 1 1 3,7 0,9 1 0,4 0,3 1,5 2 2 2 2 1 2 10 6 18 0,7 2 0,4 1,5 3,2 P 1,5 17 2 M 5 3 0,5 1,8 0,5 0,5 2,2 2 2 2 1 1 1 6 2,8 0,5 2 0,6 0,8 1,6 2 2 2 1 1 1 1,3 0,5 0,5 1,6 2,1 2 2 2 1 2 1 0,4 0,3 0,1 2,5 2 2 1 2 2 2,5 1 3 1,5 0,6 0,7 1 0,5 0,3 1,5 2 2 2 2 1 2 2 3,5 1,2 1,5 0,5 1,5 0,4 0,3 1,5 2 2 2 2 1 2 8 4,2 1,7 1 3 0,8 0,7 0,6 0,3 0,6 2,5 2 2 1 2 2 5 1,6 1,2 2,5 0,9 0,9 0,9 0,4 0,2 0,4 2,2 2 2 1 2 2 4 1 1 4 1 1 1,2 0,4 0,4 0,2 1,7 2 2 1 2 2 1 6,3 2,6 0,5 1,2 0,3 0,5 1,5 1,8 2 2 1 1 1 1,4 0,7 3,5 0,9 0,5 1 0,3 0,3 0,4 2 2 2 1 2 2 6,5 3,5 8 5 1,5 0,4 0,5 2,2 2 2 1 1 1 5,5 1 1 4 0,5 1 0,3 0,4 0,5 2,7 2 2 1 2 2 6,5 2 1 6 2 1 0,4 0,4 0,5 2,7 2 2 1 2 2 1,5 0,6 0,2 2 2 2 2 1 2 6 0,7 3 1 20 20 20 9 4,5 9 10 4,5 9 5,5 3 4 8 8 8 4,5 1,2 7,5 3,8 1 0,7 1,8 1 0,3 1 0,4 0,2 0,2 2,1 2 2 1 2 2 4,5 6 6 6 9 2 11 7 1 2,5 5 1,4 1 1 0,5 0,6 0,5 2,7 2 2 1 2 2 16 16 16 3,5 2 3 4,5 2 3 4 1,2 1,5 0,5 0,2 1,5 1,5 2 2 2 1 2 4,7 6 6 5 7 1,7 0,5 1 0,4 0,4 0,8 2 2 2 1 1 1 4 10 24 24 24 99 5,5 11 3,2 2 1,5 0,4 1 2,5 3,6 2 2 1 1 1 33 3 3 8 8 8 6,5 3 4,7 4,5 1,7 0,4 4 1,5 1,5 0,4 0,4 1,7 2,1 2 2 1 2 2 34 5 3 2 2 2 4 1 5 6 0,5 4,5 0,5 2 0,6 0,2 0,5 1,8 2 2 1 2 2 35 3 6 8 8 8 15 4 2,5 14 3 10 2,2 1 0,2 0,5 1,5 1,7 2 2 1 1 1 36 2 3,2 8 8 8 7,2 1,6 4,7 4 1 3,5 1,1 1,1 0,5 0,4 1,2 2,5 2 2 1 2 2 37 4 4,5 6 6 6 7 4 8 3,5 2 3 4 1,6 1,5 0,4 0,4 0,5 2,6 2 2 1 2 2 38 3 3 6 7 8 4 1 5 5 0,8 2 4 0,6 1 1 0,3 0,3 0,3 2,5 2 2 1 2 2 39 1 12 12 12 1 4,5 1,2 2 0,6 1 0,5 0,2 1 2,5 2 2 2 1 2 40 1 12 12 12 3,5 1,5 1,5 1 0,5 0,2 1 2 2 2 1 2 41 1 14 16 18 1,1 0,5 0,5 1,2 2 2 2 1 2 42 4 6 12 12 12 13,6 10 43 4 7 14 14 14 99 44 5 3 8 8 8 45 1 46 3 47 2 48 1 6 8 24 24 24 10 12 12 12 11,5 26 26 26 10 10 10 2,3 7 1,6 3 1 3,5 3 1,2 10,3 8,5 4,8 1,6 2 0,6 0,8 1,6 3 2 2 1 1 1 99 3 11 6,5 0,5 2,5 0,7 0,8 2,3 3 2 2 1 1 1 2 2,3 8 2 0,8 0,2 0,5 1,3 1,5 2 2 1 1 1 1,6 0,6 0,2 1,2 3,5 2 2 2 1 2 2,3 2 2 2 1 2 2 2 2 1 2 2 2 2 1 2 3 8 3,2 5 1 6,9 1 5,2 2 0,8 7 3 99 1,5 0,3 0,5 2 8,5 7,6 1,5 1,5 0,5 0,6 2 4,5 1,6 0,8 1 0,5 0,2 1 2,5 72 49 2 4 10 10 10 5 2 3 5 3 1,5 4 1,2 1 1,2 0,4 0,3 1 2,5 2 2 2 1 2 50 2 13 16 16 16 4,5 1,5 2 10 6 7 4 2 2 1 0,7 0,5 1,4 2,5 2 2 2 1 2 51 1 12 12 12 0,8 2,4 2,5 0,5 1,3 0,5 0,6 1,5 2 2 2 1 2 52 3 11 18 18 18 2 1,5 0,6 1 2,3 2 2 2 1 2 53 2 6 24 24 24 7 3 3 2 2 0,8 0,7 2 2 2 2 1 2 54 3 1,7 4 4 4 10 3 7,3 6 1,8 0,7 3,2 1,1 0,3 1 0,5 0,4 1,2 2,3 2 2 1 2 2 55 3 4,2 6 6 6 12 3,3 3 5,5 1,4 0,3 4,5 1,7 0,4 1,5 0,5 0,5 1 2,5 2 2 1 2 2 56 5 5,5 4 4 4 6,5 1,2 5,5 1 1,5 0,6 0,4 0,5 3,2 2 2 1 2 2 57 1 11 2,1 1,4 0,3 0,3 1,3 2 2 2 2 1 2 58 1 59 2 60 1 61 4 62 9 4,2 36 36 36 16 16 16 3,4 1,1 1,8 2,5 1 1 4,8 1,2 0,8 1,3 0,4 0,5 1,2 2 2 2 1 2 14 14 14 4 2 3 3 2 3 4 3 1 1,8 0,8 1 2,5 2 2 2 1 3 14 14 14 4 0,5 2,5 4 1,4 0,9 3,3 1,2 0,5 1 0,3 0,1 0,8 2 2 2 2 1 2 5 14 14 14 3,5 8 2,5 1 1,3 0,4 0,4 1,3 1,7 2 2 1 1 1 2 4 8 9 10 4 2 2 3 2 1 3 1,5 1,1 0,4 0,6 1,5 2,5 2 2 2 1 2 63 5 4 6 6 6 6 2,1 5 6,2 1,6 2 4,5 1,3 0,9 0,4 0,4 0,5 2,3 2 2 1 2 2 64 11 6 6 6 6 7 2,5 5,5 6 2 1 0,3 0,4 0,5 2,8 2 2 1 2 2 65 4 4 8 8 8 6,5 2,5 7 7 2,3 1,5 4,5 1,7 1,5 0,3 0,2 0,3 1,8 2 2 1 2 2 66 7 4 4 4 4 7 2,5 7 6 2,5 3 5 2 1,2 0,3 0,2 0,3 2,6 2 2 1 2 2 67 6 4 6 6 6 7 3,3 5,5 6 2 1,5 4,5 2,1 0,8 0,5 0,3 0,4 2,3 2 2 1 2 2 68 2 5,5 16 16 16 5 1,8 2 4 2 2 5 1,5 1,5 0,5 0,4 1,5 2,5 2 2 2 1 2 69 2 5 18 19 20 8 3 3,5 1,5 0,5 0,4 1,5 2,5 2 2 2 1 2 70 3 5 12 14 16 5,5 1,8 2,5 6 4 6 2,5 1,5 0,5 0,3 1,2 3,2 2 2 2 1 2 71 2 5 12 14 16 5,5 1,8 2,5 6 4 6 2,5 1,5 0,5 0,3 1,2 3,2 2 2 2 1 2 72 2 9 18 18 18 3 1,8 0,5 2,5 1,4 1,1 0,3 0,1 1 2 2 2 2 1 2 73 1 16 16 16 2,5 6,5 4 99 6 2 1,5 0,7 0,3 1 3 2 2 2 1 2 74 4 5 12 12 12 7 14 6 4 9 2,6 0,8 0,4 0,4 1,5 2,2 2 2 1 1 1 75 5 3,5 10 10 10 2,5 10 1,7 99 7,5 2 1,5 0,7 1,2 1,8 3,1 2 2 1 2 2 76 2 8 26 26 26 9 5 1,5 7 3 1,6 0,7 0,4 1,4 2,6 2 2 2 1 2 77 1 12 12 12 5 3,5 4 7 2,8 1,2 6 3 1,5 0,6 0,2 1,6 2 2 2 2 1 2 78 5 2,8 23 23 23 2,6 0,8 3 3,2 1,1 1 2,2 1 0,5 0,8 0,3 0,3 0,2 2 2 1 2 2 79 3 3 6 6 6 7,2 1 6 6 1,1 2,5 6,5 0,9 1 1 1,2 0,4 0,5 2 2 2 1 2 2 80 4 5,5 6 6 6 5 1,5 5 4 1,5 1 4 2 1 0,6 0,3 1 2,5 2 2 1 2 2 81 2 2,5 6 6 6 9,5 1,6 6 7,2 1,4 1,5 3,5 0,6 0,4 1 0,3 0,3 0,2 2,3 2 2 1 2 3 82 5 5 6 6 6 5 2 4,5 4,8 2 1 5 1,8 0,6 1,5 0,5 0,5 0,7 1,6 2 2 1 2 2 83 3 7,5 8 8 8 9,5 2 3 0,5 1,4 1 8 1,1 0,5 1,2 0,5 1,5 1,7 2 2 1 2 2 84 4 4,2 6 6 6 7,3 1,9 5 5 1,1 0,7 3,8 1,1 0,2 2 2 1 2 2 85 5 4,5 16 16 16 7,3 1,7 7 6 2 1 4 1,5 0,5 86 3 3,5 6 6 6 8 3 6 5,5 2 2 87 4 5 4 5 6 7 3,5 7,5 6,5 3 88 5 2 3 3 3 4,5 0,5 5 5,5 1,2 89 5 3,5 6 6 6 7,5 3 7 5 2 90 5 5 6 6 6 7 91 4 2,5 6 6 6 5 2,5 4 92 4 4,5 6 6 6 5,8 2,1 5 93 4 4,3 36 36 36 94 5 6 6 8 95 1 24 24 96 1 97 1 98 4 99 2 100 101 4 7 9 5,5 5 2,4 1,5 0,5 0,5 0,4 0,5 2,5 2 2 1 2 2 1,2 1,1 0,3 0,4 0,2 2,6 2 2 1 2 2 5 2,5 1,5 0,5 0,5 1,2 3 2 2 1 2 2 4 1 1,2 0,6 0,4 0,5 3 2 2 1 2 2 2 3 1,5 1,3 0,4 0,3 0,7 2,7 2 2 1 2 2 1,4 1,3 6 1 0,5 1 0,4 0,5 0,7 2,3 2 2 1 2 2 7 3 1,5 4,5 2 0,7 1,2 0,6 0,3 0,7 1,5 2 2 1 2 2 4 1 1 3,5 0,9 1 0,3 0,4 0,6 2,3 2 2 1 2 2 3,2 5,5 1 1,1 0,3 0,3 1 1,1 1 1 1 1 1 2 99 4 1,5 1,2 0,3 0,4 0,5 3 2 2 1 2 2 24 5 2 1,5 3 1,7 2 0,6 0,2 1,7 3 2 2 2 1 2 16 16 16 5 2 1,5 4 1,6 2 0,8 0,2 1,7 2,8 2 2 2 1 2 18 18 18 5 2,3 1,5 3,5 2,2 1,7 0,8 0,2 1,8 2,5 2 2 2 1 2 3 8 8 6,2 2,4 2 2,5 0,7 0,5 0,9 0,4 0,2 0,4 2,1 2 2 1 2 2 3 12 12 12 4,7 1,1 1 4,2 1,4 0,6 1,2 0,5 0,7 1,3 2,5 2 2 1 2 2 2 4,5 14 14 14 4 6,5 4 1 0,5 0,5 1,5 3 2 2 1 1 2 2 5 8 8 2 4,5 1,5 1,5 0,5 0,2 1 2 2 2 2 1 2 8 5 0,5 6,5 8 3 1 1 8,5 9 1 9 8 3 3 5,5 3 73 102 2 103 1 104 2 105 106 7 16 18 20 3 1,2 2 5 2,5 4,5 2,2 1,3 0,4 0,2 1,2 2,6 2 2 2 1 2 12 14 16 9 2,5 8 8 3,5 8 3 1,8 0,7 0,3 1,5 4 2 2 2 1 2 4,5 14 14 14 4,5 1,5 2 1 4 2,4 1 0,3 0,3 1 2,5 2 2 2 1 2 2 4 18 18 18 4,5 1,4 1,5 4,7 2 1,5 2 1 1 0,5 0,3 1,4 2 2 2 2 1 2 2 2,5 10 10 10 3,5 2 2 3 2 4 3 1,5 1,5 0,4 0,5 1,6 3 2 2 2 1 2 107 2 4,5 8 10 4 2 1,5 3,5 2 2 4 2 1,5 0,5 0,9 1,8 3 2 2 2 1 2 108 1 24 24 24 8 2 7 6 2 2 1,2 0,4 0,6 1,5 2,3 2 2 2 1 2 109 1 10 10 10 7 2,5 3 3,5 3,5 4 6 3,5 2 1,2 0,4 0,5 1,6 99 2 2 2 1 3 110 2 5,5 10 11 12 7,5 2 3 3,5 3,5 2 5 1,3 1 1,5 0,5 0,6 1,8 3 2 2 2 1 3 111 2 3,5 12 12 12 6 2,5 1,5 2,5 2,5 1 3,5 1,2 1,6 0,6 0,8 1,5 2,6 2 2 2 1 3 112 2 7,5 12 12 12 1 6,2 1 1 0,5 0,5 1 2 2 2 2 1 2 113 2 13 16 16 16 5 1,5 2 4,5 2,1 1,5 5,5 1,1 1 0,3 0,1 0,8 2 2 2 2 1 2 114 2 9 24 24 24 6,5 1,4 3,2 7 2,8 1 8 1,7 1,3 0,6 0,3 1 2,5 2 2 2 1 2 115 2 4,5 16 16 16 5,5 2 4,5 4 2,5 2 4 2 1,5 0,6 0,3 1 3 2 2 2 1 2 116 2 4,5 6 6 6 3 2,5 4 2,4 1,8 4 1,6 1,6 0,5 0,7 1,6 1,5 2 2 2 1 3 117 1 14 14 14 5 2,3 2 1,3 0,5 0,5 1,7 2,5 2 2 2 1 2 118 6 4 23 18 16 8 4 7 9 4 8 5 2,5 2 0,5 0,5 1,5 2 2 2 1 2 2 119 3 3,5 6 6 6 10 5 15 6 2 2 4 1,5 1,5 0,6 0,4 0,3 2,8 2 2 1 2 2 120 4 4 6 6 6 5,5 2,2 6 5 1,5 4 1,3 1,5 0,4 0,3 0,4 2,5 2 2 1 2 3 121 4 4 6 6 6 4,5 1,6 5,5 4 2 2,5 3,5 1,2 1,2 0,3 0,4 0,5 2,5 2 2 1 2 2 122 3 5,5 6 6 6 11 1 6 3,2 1,5 1 5 0,9 1,3 0,5 0,7 1 2,5 2 2 1 2 3 123 4 4,5 6 6 6 5 1 6 6 2 5 2 1,4 0,5 0,8 1,2 2,8 2 2 1 2 2 124 7 3,5 6 6 6 6,5 3 5 6 2,5 1,5 3,5 1,8 1,5 0,5 0,5 0,4 2,8 2 2 1 2 2 125 5 4,5 6 6 6 8 5 7 8 3 2 4,5 3 1,3 0,4 0,5 99 2,6 2 2 1 2 2 126 3 4 10 10 10 3,5 6 2 2 6 2 1 0,4 0,5 1 2 2 2 1 1 1 127 4 4,5 8 8 8 7,7 1,5 8 7,5 1 4,5 1,1 0,7 0,4 0,4 1 2,2 2 2 1 2 2 128 3 1,5 6 6 6 3,5 1,5 2 3,5 0,8 1,5 2,5 1 1 0,4 0,2 0,2 1 2 2 1 2 2 129 4 3,5 8 8 8 12 4 11,5 8 6 1 3,5 1,2 0,3 1 0,5 0,3 0,3 2 2 2 1 2 2 130 2 4,5 14 14 14 5,5 3,5 3 2 4 2 2 1,8 0,8 0,8 1,7 2 2 2 1 3 131 1 22 22 22 3,5 1,6 1 1 0,5 0,4 1,3 1,9 2 2 2 1 2 132 3 4 10 10 10 5 1,5 2 2 1,5 2,5 4 2 1,6 0,6 0,6 1,5 99 2 2 2 1 133 2 4 8 10 6 3,2 2,5 4 2,5 3 4,5 2 1,3 0,4 0,6 2 2,5 2 2 2 1 2 134 1 24 24 24 8 3,4 5 0,5 1 0,4 0,5 1 2 2 2 1 2 135 5 5,5 8 8 12 4 2,5 9 3 1,5 7 1 1 0,8 0,3 0,3 1 2 2 1 1 1 136 2 2,5 10 10 10 2,5 1,5 1,5 2 1 1 3 1,5 0,5 1 0,4 0,5 1,2 2 2 2 1 2 137 5 2 8 8 8 1,7 1 3 3,2 1,6 0,5 2,5 1,4 0,5 0,8 0,3 0,2 0,3 2 2 2 1 2 2 138 6 3,5 6 6 6 12 6 2,5 8,5 3 2 8 3 0,8 0,2 0,5 1,6 1,5 2 2 1 1 1 139 4 4,5 6 6 6 5 2 5 5 3 3 1 1,2 0,4 0,4 0,4 2,4 2 2 1 2 2 140 4 3,5 6 6 6 6 2 5 5 1,8 1 5 1,2 1 0,3 0,2 0,5 2,6 2 2 1 2 2 141 5 4 10 10 10 7 3,5 7 3 1,5 0,4 5 3 0,6 1,3 0,4 0,3 99 1,8 2 2 1 2 2 142 3 8 16 16 16 12 3 5 9 2,4 1,5 1,8 0,5 0,8 2,4 4 2 2 1 1 5 143 4 6 28 28 28 12 3 4 10 3 2 1,8 0,4 0,5 2,5 4 2 2 1 1 1 144 5 4,5 6 6 6 10 2,5 99 7 1,4 1,3 0,4 0,2 0,7 2,4 2 2 1 1 1 145 3 2,5 8 8 8 3,5 0,9 0,5 2,4 1,1 0,3 1 0,3 0,4 0,6 3,3 2 2 1 2 2 146 3 4,5 18 18 18 0,3 1,5 0,3 0,5 1,5 1,5 2 2 1 1 2 147 2 7 10 10 10 4,5 1 1,5 3,5 2,2 2 22 1,2 1,3 1 0,5 0,2 1 2 2 2 1 2 148 1 12 12 12 3,6 1,4 1 3,7 2,2 1,3 3,9 1,4 0,8 1 0,5 0,2 1 2 2 2 1 2 149 5 2 12 12 12 13 4,5 9 8 3 2 5,5 1,5 1 0,6 0,4 99 0,5 2 2 1 2 2 150 2 6 16 16 16 5 2 3 10 6,5 4 7 5 1,5 2 0,4 0,5 1,7 3,5 2 2 2 1 2 151 5 6,5 14 14 14 7 1,2 1,5 8 1,4 0,4 5,5 1,5 99 1,5 0,6 1,5 2,5 0,9 2 2 1 2 2 152 3 4 4 4 4 8,5 2,2 6,5 6 1 0,5 3,5 0,6 0,6 1 0,4 0,2 0,4 2 2 2 1 2 3 153 5 3,5 6 6 6 8,5 3,3 7 4,5 1,2 1 3,4 1 0,6 1 0,4 0,3 0,2 2,5 2 2 1 2 2 154 4 3 10 6 6 16 5 8 10 3,2 2,2 2,4 0,8 0,6 1 0,4 0,3 0,8 2 2 1 2 2 9 6 9 8 2 99 4,5 8 1 3,5 0,7 0,8 1,4 1,2 2 4 1 0,5 1,5 1,3 2,2 74 155 5 2,5 6 6 6 6,5 1,5 4 6,5 3 4 1,8 1,1 0,5 0,9 1,2 2,7 2 2 1 2 2 156 5 5,5 6 6 6 8 2,5 6 5,5 1,5 1 5 1,5 1,5 0,3 0,2 0,3 2,5 2 2 1 2 2 157 4 3 8 8 8 4 2,2 2,2 4 1,5 0,5 2,4 1,2 0,7 1 0,3 0,2 0,1 2,5 2 2 1 2 2 158 2 4 6 6 6 2,5 1 0,5 5 1,5 0,3 3,5 1,2 0,5 1,5 0,4 0,3 0,2 1,8 2 2 1 2 2 159 6 1,5 8 8 8 4,2 2,2 4,5 4 1,6 1 3,5 1,2 0,5 1 0,7 0,3 0,5 1,6 2 2 1 2 2 160 5 2 6 6 6 11 4 9 8 3 2,5 3,5 1,2 0,5 1 0,5 0,3 0,3 1,5 2 2 1 2 2 161 4 6 14 14 14 14 4 12 9 4,5 5 8 3 2 1,5 0,5 0,6 2 2,5 2 2 1 1 5 162 5 3,5 6 6 6 8 3 4 6,5 2 0,8 0,3 0,4 1 1,5 2 2 1 1 1 163 4 4,5 5 5 5 12 6 10 5 4,5 7 2 0,8 0,3 0,6 1,4 1 2 2 1 1 1 164 5 2 8 8 8 7 8 5,5 2,5 1 2,5 1 1,2 0,5 0,3 0,5 1,5 2 2 1 2 2 165 3 14,5 32 32 32 2,5 12 7 1,5 0,5 0,5 2 2,5 2 2 1 1 2 166 6 20 24 24 24 13 15 9 3,5 1,7 0,7 0,3 2 3 2 2 1 1 2 167 6 19 24 24 24 18 15 8 3,5 1,3 0,6 0,3 2 2 2 2 1 1 2 168 4 3 8 8 169 4 3,8 30 30 30 170 3 2 4 4 171 4 2 172 5 5 173 3 174 6 7,5 2 2 175 5 9,7 6 176 4 4 177 5 3 178 7 179 8 8 4 8 10 2,6 2 0,4 7 8,5 1 2 5,5 0,7 1,5 0,6 0,2 1 1,8 2 2 1 2 2 9 5,5 2,8 0,8 3,2 1,2 1 0,3 0,3 0,9 1,7 1 1 1 1 1 0,6 0,2 0,1 1 2 2 1 1 4 0,2 2 2 1 2 2 2 2 1 1 1 2 2 1 2 2 2 2 1 2 2 25 5 15 12 2,5 2 4,5 1 8,5 4 9 10 2,5 4,5 5 2 0,8 7 3 1,2 7,2 2,2 1,2 2,5 1,7 0,8 0,4 8 1 0,3 0,3 1,6 2 7 2,5 9 7,8 3 2 9,1 1,1 6 11,3 0,7 7,1 0,5 1,5 0,5 0,4 6 5 8,3 1,1 3 10,5 0,7 8,3 0,6 1,5 0,4 0,3 0,3 2,8 2 2 1 2 2 4 6 5 4,6 2,2 8 2,5 1,2 3 1,3 1,2 1 0,5 0,3 0,5 2,4 2 2 1 2 2 6 6 6 4 1,5 3 2 0,6 0,5 2 0,6 0,4 0,3 2 2 2 1 2 2 5 6 6 5 11 1,9 11 8,4 1 2,2 8,7 1,1 99 1,3 1,7 0,5 0,5 3 2 2 1 2 2 6 5,5 5 5 5 9,9 3,5 9 9,8 2,8 3 5 2 1,5 1,7 0,6 0,7 0,5 2,8 2 2 1 2 2 180 7 5 6 6 5 11 1,9 11 8,4 1 2,2 8,7 1,1 1,3 1,7 0,5 0,5 3 2 2 1 2 2 181 7 3,5 2 2 2 11 1,8 7 4,5 0,9 3 0,5 2 0,8 0,3 2,7 2 2 1 2 2 182 4 3 5 6 7 7,3 3,1 6,5 4,3 2,9 1 3,3 2,3 0,6 1,7 0,5 0,5 0,8 2,5 2 2 1 2 2 183 4 6 2 2 2 11 2,7 8 6,7 2,4 1,2 3,3 1,4 0,8 1,6 0,5 0,5 0,3 2,7 2 2 1 2 2 184 1 12 12 12 4 1,4 1 3,7 2,2 1,3 3,9 1,4 0,8 1 0,5 0,2 1 2,2 2 2 2 1 2 185 2 2,5 6 6 6 8,5 0,8 6 7,5 0,6 2 3 0,4 0,5 1 0,5 0,4 0,6 1,5 2 2 1 2 2 186 2 7 7 7 7 3,5 1 2 3,6 1,2 0,8 3 1 1 0,4 0,1 0,9 2 2 2 1 2 187 4 4,5 7 7 5 2 3 6 2,5 2 0,3 0,5 1,8 2 2 2 1 3 188 1 5 2 1,5 0,7 0,5 1,5 2 2 2 1 2 189 6 1,2 0,4 0,4 0,5 2 2 1 2 2 190 2 2 2 1 2 0,8 7 4 3,3 4 30 30 30 6 3 4 6 6 6 3,5 3,5 9 6 3 1 4,5 2 2 5 16 16 16 3,5 1,5 2,5 2,5 2 2 4 1,6 0,6 1,2 0,4 0,7 1,5 191 2 4,5 10 10 10 5 1 3 4 2 1 4 1,5 1 1,2 0,4 0,4 1,2 1,5 2 2 2 1 2 192 2 3,5 8 8 9,5 2,2 1 6,5 1 5 1,2 0,3 0,4 1 1,5 2 2 1 1 1 193 2 9 12 12 6,5 0,4 1 3,5 0,3 1 0,4 0,3 0,5 2,4 2 2 1 2 2 6 8 12 6 0,4 4 2 0,3 3 75 Ek 2.Tüm Örneklerin ISSR Ürünlerinin Agaroz Jeldeki Görüntüleri •UBC 810 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M: Marker ; Vivantis 3000 bç) 76 •UBC 810 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M: Marker ; Vivantis 3000 bç) 77 •UBC 811 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 78 •UBC 811 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 79 •UBC 814 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 80 UBC 814 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 81 •UBC 815 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 82 •UBC 815 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 83 •UBC 815 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 84 •UBC 818 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 85 UBC 818 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 86 UBC 822 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 87 UBC 822 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 88 •UBC 823 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 89 UBC 823 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 90 •UBC 824 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 91 •UBC 824 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 92 •UBC 826 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker; Vivantis 3000 bç) 93 •UBC 826 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker; Vivantis 3000 bç) 94 •UBC 827 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 95 •UBC 827 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 96 •UBC 835 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 97 •UBC 835 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 98 •UBC 836 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 99 •UBC 836 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 100 •UBC 840 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 101 •UBC 840 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 102 •UBC 841 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 103 •UBC 841 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 104 •UBC 842 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 105 •UBC 842 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 106 •UBC 844 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 107 •UBC 844 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 108 •UBC 848 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (M:Marker ; Vivantis 3000 bç) 109 •UBC 848 kodlu ISSR primeri ile 193 Phlomis örneğinin PCR amplifikasyonunun agaroz jelindeki görüntüsü (Devamı) (M:Marker ; Vivantis 3000 bç) 110 ÖZGEÇMİŞ Kişisel Bilgiler Adı, Soyadı: Özge FIRAT Uyruğu: Türkiye (TC) Doğum Tarihi ve Yeri: 14 Temmuz 1985,Ankara Medeni Durumu: Evli Tel: +905317766047 email: biyoozge@hotmail.com Yazışma Adresi: Yıldırım Beyazıt mah. Sırasöğütler caddesi beyazıt apt no:7 KAYSERİ Eğitim Derece Kurum Mezuniyet Tarihi Lisans EÜ FEF Biyoloji 2009 Lise Kocatepe Mimar Kemal Lisesi 2003 İş Deneyimleri Yıl Kurum Görev 2012 Gelişim dershanesi Biyoloji öğretmeni 2014 MEB Sözleşmeli Biyoloji öğretmeni Yabancı Dil İngilizce